Nat Commun:基于纳米孔测序的mRNA疫苗质量分析新方法,兼顾效率与准确性!
时间:2023-10-31 14:39:55 热度:37.1℃ 作者:网络
2023年的诺贝尔生理学或医学奖颁给了mRNA疫苗技术的两位“功臣”:卡塔林·卡里科和德鲁·维斯曼,以表彰他们在mRNA研究领域的突破性发现,助力疫苗开发达到前所未有的速度。
mRNA疫苗在新冠肺炎大流行期间已经展现了较高的安全性和有效性。mRNA疫苗的应用范围包括传染性疾病、癌症、自身免疫性疾病等。毫无疑问,mRNA 疫苗和相关疗法具有广阔的前景。生物制造业目前可以支持数十亿剂疫苗的生产,同时在质量和安全性方面都有足够的保证,所以mRNA疫苗的成功在一定程度上是依赖于生物制造业的进步。
在mRNA生产过程中,必须对质量进行严格分析,测定其完整性,检测会降低疫苗有效性并引发副作用的污染物。目前,mRNA的分析涉及多种技术(例如RT-qPCR、毛细管电泳和凝胶电泳、RP-HPLC、IP-RP-HPLC和免疫印迹),这些技术的使用既繁琐又昂贵,而且通常无法灵敏地检测关键的mRNA质量特征,因此亟需优化mRNA的分析过程。
近日,来自澳大利亚昆士兰大学的研究团队在Nature Communications发表了题为“mRNA vaccine quality analysis using RNA sequencing”的研究,建立了一种使用纳米孔测序分析mRNA疫苗和疗法的简化方法——VAX-seq。与行业内其他的标准技术相比,VAX-seq可以全面检测关键的mRNA疫苗质量特征,包括序列、长度、完整性和纯度。研究人员还描述了如何使用RNA测序数据分析mRNA化学,包括核苷修饰的检测,并提供了数据分析软件工具Mana来自动报告mRNA和质粒模板的质量和完整性。

文章发表在Nature Communications
主要研究内容
目前,RNA测序技术已经广泛用于细胞基因表达的分析,可以用于检测mRNA的序列同一性和定量丰度。虽然RNA测序可以获得内源性mRNA的特征,但尚未用于mRNA药物的分析和制造。因此,研究团队评估了短读长测序和长读长测序在mRNA疫苗和治疗分析中的应用,并开发了VAX-seq使用纳米孔cDNA测序分析mRNA疫苗。(图1)为了支持VAX测序数据的分析,研究团队还开发了一个软件工具Mana将比对的NGS文库作为输入,并报告质粒和mRNA长度、序列同一性和纯度。
mRNA疫苗和疗法是通过快速、无细胞的体外转录制备的。mRNA制造首先是进行质粒DNA(pDNA)模板的制备和线性化,该模板通过RNA聚合酶在体外转录并合成mRNA,其中通常包含5′ cap 和模板编码的3′poly(A)尾(图1)。随后,还必须纯化mRNA中的污染物,包括反义和双链RNA。这些污染物会引发先天免疫反应,抑制细胞翻译并诱导副作用。
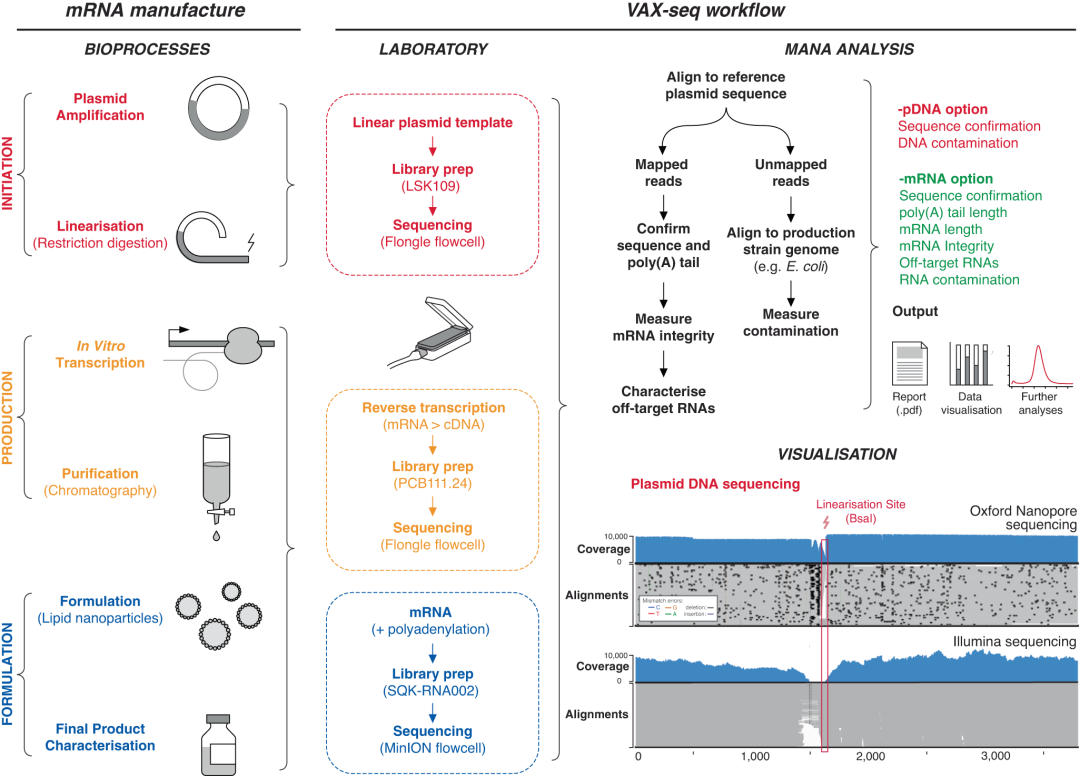
图1. mRNA疫苗生产和VAX-seq工作流程
1.mRNA序列一致性和完整性分析
mRNA疫苗制备的第二步,是将线性化质粒作为合成mRNA的体外转录模板,将其从剩余DNA、RNA和蛋白质中纯化出来,并且需要进一步检测以确认序列同一性、长度、完整性。研究人员使用长读长cDNA测序(SQK-PCS111)分析了纯化的mRNA,reads经过处理并与参考质粒序列比对,通过Mana软件进行分析,确定了mRNA的一致序列(consensus sequence)和长度。结果显示,VAX-seq可以检测mRNA疫苗的长度,并对mRNA样本完整性进行定量。对于单个reads,测序平均错误率为5.0%,poly(A)尾部的缺失率较高(13%)(图2a-c)。
据文章介绍,VAX-seq是将逆转录酶引物锚定在poly(A)尾的3’末端,使得完整的poly(A)尾能够被检测其长度(图2d)。但由于缺失误差,长度平均低估了约11.2%(图2d)。这些误差是系统性的,为了校正这些缺失错误,研究人员使用tailfindr软件评估了原始数据,将reads特异性核苷酸易位率归一化。对于eGFP mRNA,预期的126nt相比,tailfindr准确估计poly(A)尾部长度为126.04nt(图2e)。
此外,由于使用了纳米孔测序,VAX-seq可以检测mRNA疫苗的长度,并提供mRNA完整性的定量检测。研究团队通过分析三个重复文库,证明了VAX-序列工作流程测量mRNA完整性的可重复性。以上结果表明,纳米孔测序能够提供任何mRNA疫苗样品在制造、储存或交付过程中的完整性定量分析。
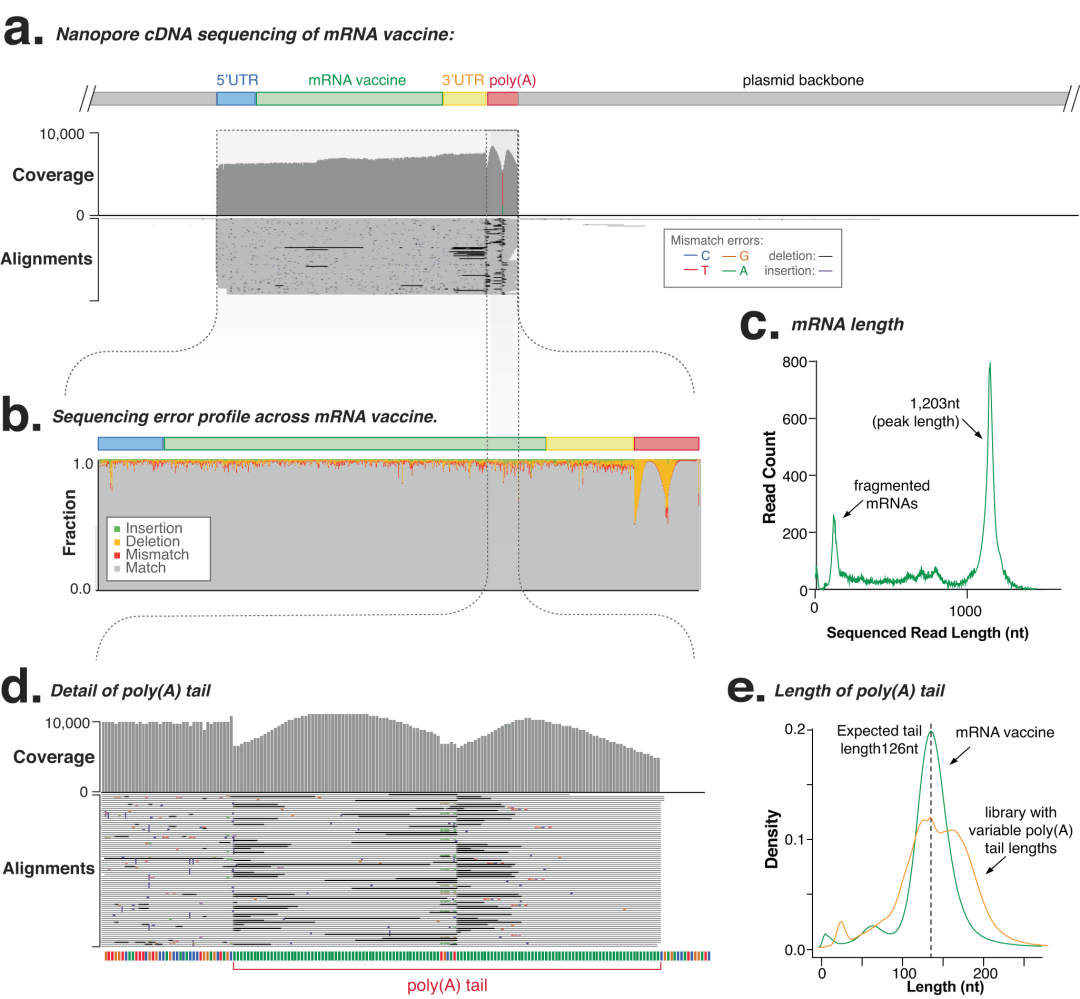
图2. 使用纳米孔测序(PCS111)分析参考eGFP mRNA疫苗
为了进行比较,研究人员还使用短读长cDNA测序分析了合成mRNA,并使用优化的Mana工作流程分析了文库,短读长测序确认了mRNA序列的一致性和准确性(图3a,b)。但poly(A)尾部的短reads的错位导致了许多错误和较差的一致准确性(consensus accuracy),此外,短读长测序显示覆盖度较差且不均匀,无法对mRNA长度和完整性进行分析。以上结果突出了使用短读长测序分析低复杂性序列的挑战性。
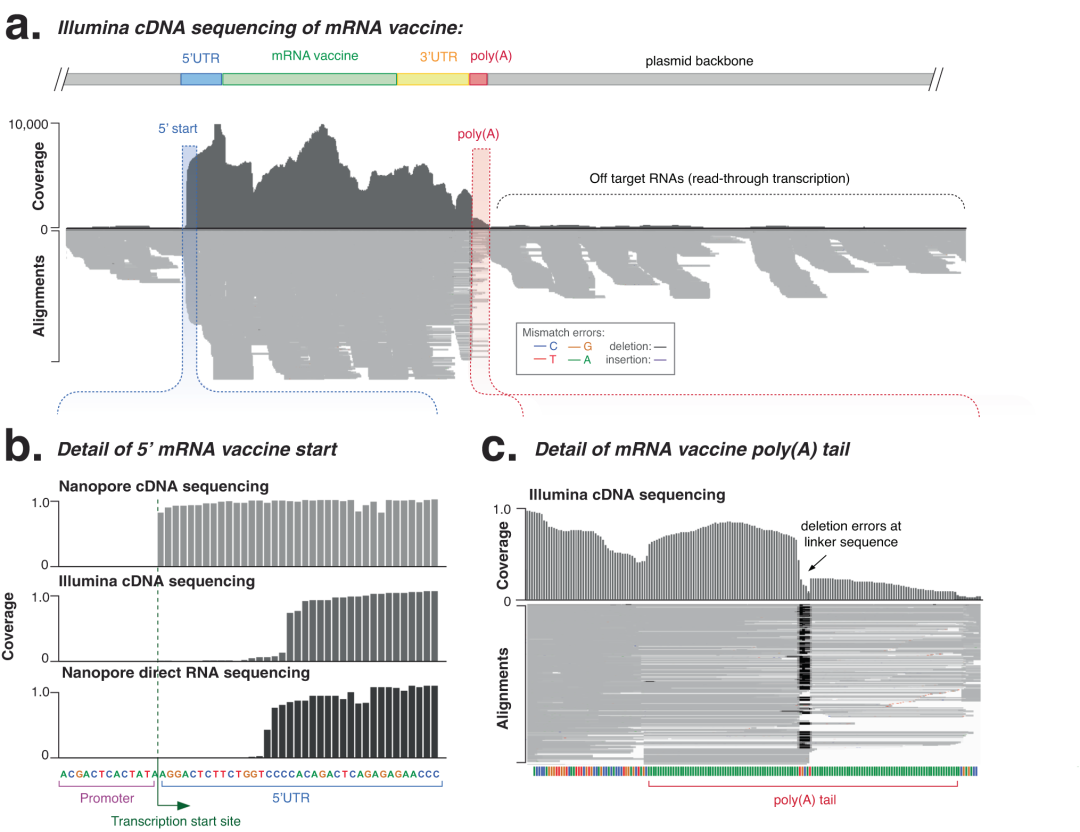
图3. 使用短读长测序(TruSeq)分析参考eGFP mRNA疫苗
2.表征脱靶RNA
体外转录产生的脱靶RNA可引发先天免疫反应,因此必须将其去除以确保mRNA疫苗的安全性和有效性。研究团队使用VAX-seq分析了cDNA文库中的脱靶RNA的特征,大多数序列(92.7%)与靶mRNA产物比对,只有少数reads(0.01%)被检测到污染,剩余的(7.3%)RNA包含不同的脱靶RNA。(图4e)
为了比较,研究团队也用短读长cDNA测序分析了合成的mRNA。短读长测序也检测到类似的脱靶RNA,包括上游(1.4%)和下游(5.7%)序列。短读长链定向分析还可以灵敏地检测到约0.6%的反义RNA转录本。
3.mRNA疫苗的直接RNA测序
纳米孔测序能够直接测序合成的mRNA,在文库制备过程中无需进行逆转录和扩增,并且可以直接分析单个mRNA疫苗分子中的核苷修饰。从直接RNA测序(SQK-RNA002)结果显示,与匹配的cDNA测序文库相比,直接RNA测序文库的产量较低,并且它们目前不能被多路复用,如果在mRNA制造过程中进行大规模测序,应考虑这一点。mRNA疫苗质量分析(poly(A)尾)显示,虽然直接RNA测序提供了足够质量的一致序列,但其平均错误率(7.78%)高于匹配的cDNA测序(图4b)。
详细的分析是开发和制造mRNA疫苗和疗法的关键。VAX-seq能够使用纳米孔cDNA测序分析mRNA疫苗。研究人员设计并制造了参考eGFP mRNA,用于验证VAX-seq方案。eGFP mRNA疫苗包含以下组成部分(按5’至3’的顺序):(i)用于体外转录的T7 CleanCap启动子,(ii)α-球蛋白5’UTR,(iii)增强型绿色荧光蛋白(eGFP)开放阅读框,(iv)AES-mtRNR1 3’UTR,和(vi)用于模板线性化的限制性酶(BsaI)消化位点(图4e)。
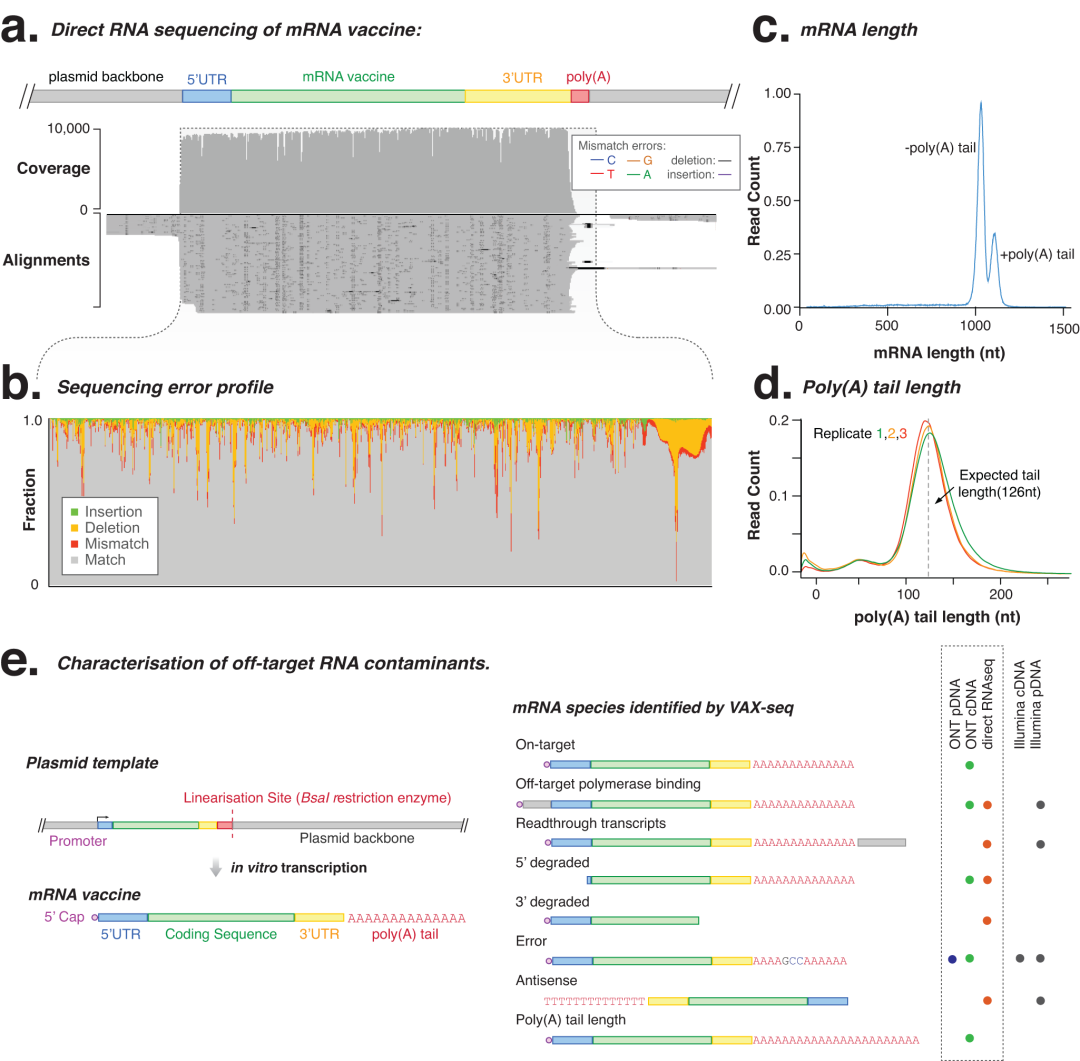
图4. 参考eGFP mRNA疫苗的直接RNA测序
通过分析修饰核苷对mRNA质量属性的影响,cDNA和直接RNA测序都表明,修饰的mRNA疫苗包含的截短转录物更多,特别是长度低于500nt的mRNA(图5c)。
结 语
综上所述,研究团队基于纳米孔测序开发的VAX-seq,能够灵敏、定量地检测关键的mRNA质量特征,包括序列一致性,完整性和污染物。VAX-seq可以在不同的制造步骤检测mRNA的质量,从最初的质粒制备到最终产品的表征,提供单一全面的综合分析。此外,VAX-seq只需要很少的mRNA作为输入,能够支持大批量疫苗批次的大规模验证。
参考资料:
Gunter, H.M., Idrisoglu, S., Singh, S. et al. mRNA vaccine quality analysis using RNA sequencing. Nat Commun 14, 5663 (2023). https://doi.org/10.1038/s41467-023-41354-y


