Nat Med:Paige、微软携手发表迄今最大的计算病理学基础模型Virchow,为AI癌症病理学树立新标杆
时间:2024-08-13 19:01:56 热度:37.1℃ 作者:网络
组织样本的病理分析对于癌症的诊断和治疗至关重要。近年来,数字化全切片图像(WSIs)正逐步取代光学显微镜传统组织病理学检查。WSIs是计算病理学工作的基础,有助于应用AI进行疾病诊断、表征。在这一领域,FDA于2021年9月批准了首个AI病理系统,用于解读常规WSIs以获取以前未知的结果,如预后和治疗反应。近期计算机视觉的进步促使研究者探索从常规WSIs中获取更多医疗洞见,以提升H&E全切片图像的实用性并减少对特定昂贵测试的依赖。
计算机视觉模型的性能提升得益于基础模型的创建。基础模型是一种建立在海量数据集上的自我监督算法,极大地增强了AI在图像分析领域的应用。这些模型的显著优势在于它们无需人工标注,这提供了泛化能力和训练数据效率。要做到这一点,需要大规模数据集和强大的计算资源。
近日,AI病理学解决方案提供商Paige公司联合微软发表了合作开发的顶级AI癌症病理基础模型——Virchow,展示了其如何在超过100万个WSIs上进行训练(同类数据集中规模最大),从而具备对病理学图像中观察到的各种模式进行前所未有的建模能力。数据显示,Virchow在生物标志物预测、细胞识别和泛癌检测方面具有一流的性能。该研究以“A foundation model for clinical-grade computational pathology and rare cancers detection”为题发表在Nature Medicine。

文章发表在Narure Medicine
Virchow以现代病理学先驱Rudolf Virchow的名字命名,利用MSK癌症中心约10万名患者的150万张H&E染色WSIs进行训练,数据规模是以往数据集的4-10倍,像素高3000倍。训练数据涵盖17种高级组织的癌性和良性组织,通过活检(63%)和切除(37%)收集。Virchow是一个具有6.32亿参数的ViT模型,采用DINO v.2算法进行自监督训练,该算法利用组织切片的全局和局部区域来学习生成WSIs切片的嵌入,并用于训练各种下游预测任务(图1)。
目前,Virchow模型嵌入在两类切片级计算病理学应用上进行了评估:泛癌检测(“Virchow实现临床级泛癌检测”)以及生物标志物预测(“常规成像中的生物标志物检测避免了额外测试”)。这些任务需要训练一个弱监督聚合模型,以将切片嵌入整合为切片级预测。研究团队还进行了一系列切片级线性探测基准测试,以直接评估单个组织切片上的嵌入(“切片级基准测试和定性分析证明其泛化能力”)。
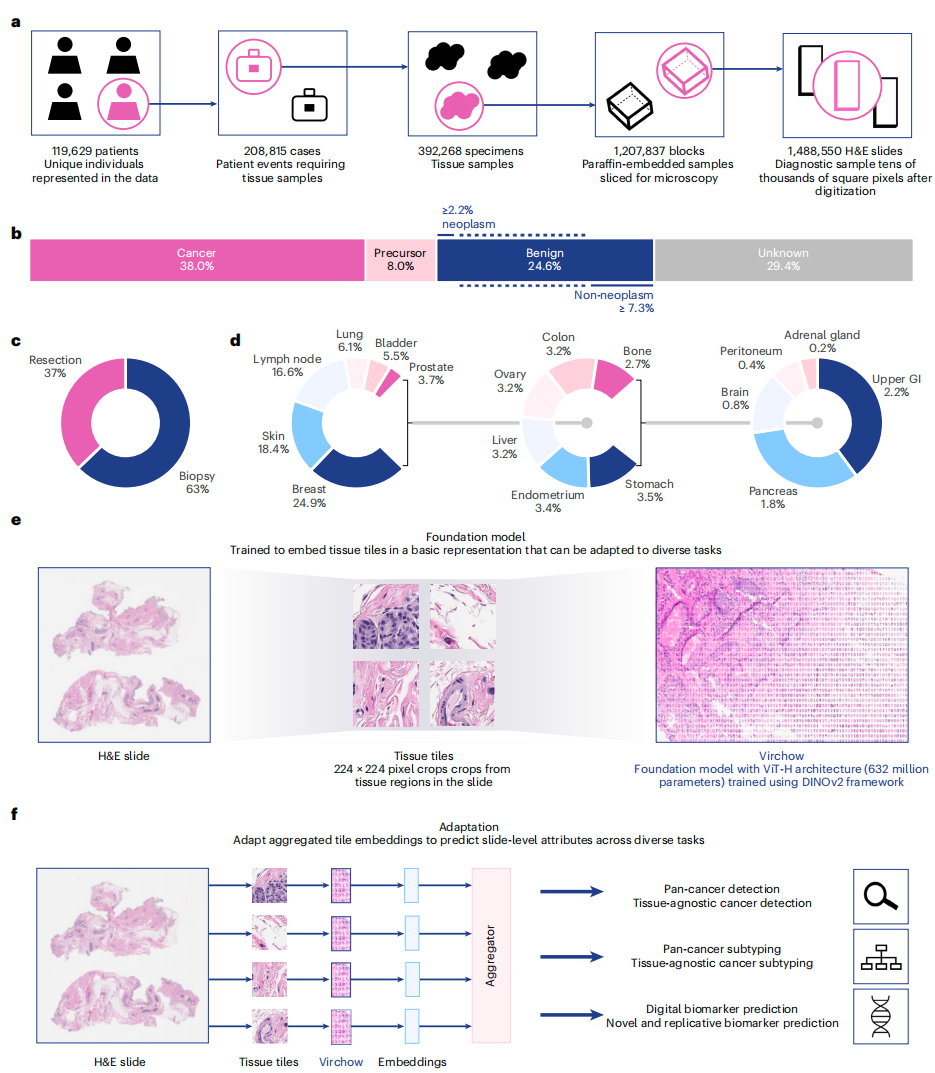
图1.研究概述
Virchow实现泛癌检测
该研究的关键目标是开发一种单一模型来检测各种组织中的癌症,包括罕见癌症。泛癌检测模型使用Virchow嵌入作为输入来推断癌症的存在,对来自MSK和全球多个机构的切片进行评估,报告了9种常见和7种罕见癌症类型的分层性能。研究还评估了由Virchow、UNI、Phikon和CTransPath模型生成的嵌入,所有嵌入均使用样本级别的标签进行泛癌聚合器的训练。
数据显示,Virchow嵌入在所有癌症类型检测中表现最佳(图 2a),使用Virchow嵌入的泛癌模型AUC为0.950,使用UNI嵌入为0.940,使用Phikon嵌入为0.932,使用CTransPath嵌入为0.907;与其他嵌入相比,Virchow模型的AUC更高,在95%灵敏度下,使用Virchow嵌入的泛癌检测模型特异性更高且训练数据更少(图2b)。同时,Virchow嵌入的罕见癌症检测有较好表现(AUC为0.937),对各罕见癌症的性能不均,在宫颈和骨癌检测上仍具挑战,但Virchow嵌入在这两种癌症检测上的AUC高于其他嵌入(图2c,d)。
研究人员采用非MSK癌症中心的数据评估Virchow嵌入对来自域外数据(OOD数据)的鲁棒性(图 2e,f)。结果显示,使用Virchow嵌入的泛癌模型在不同数据集上显示出一致的AUC差异,表明其具有良好的泛化能力。此外,Virchow嵌入在未训练过的组织(如宫颈、睾丸和头颈部)数据上仍然优于其他竞争模型。总体而言,泛癌症检测在包括罕见癌症在内的癌症类型以及OOD数据上具有泛化能力。基于不同基础模型嵌入的泛癌检测性能随基础模型的大小和训练数据量而扩展,但存在收益递减的趋(图 2g)。虽然Virchow、UNI、Phikon和CTransPath在训练数据集、模型架构和优化策略上有所不同,但所有模型均是基于变换器,且性能扩展分析显示了相似性。
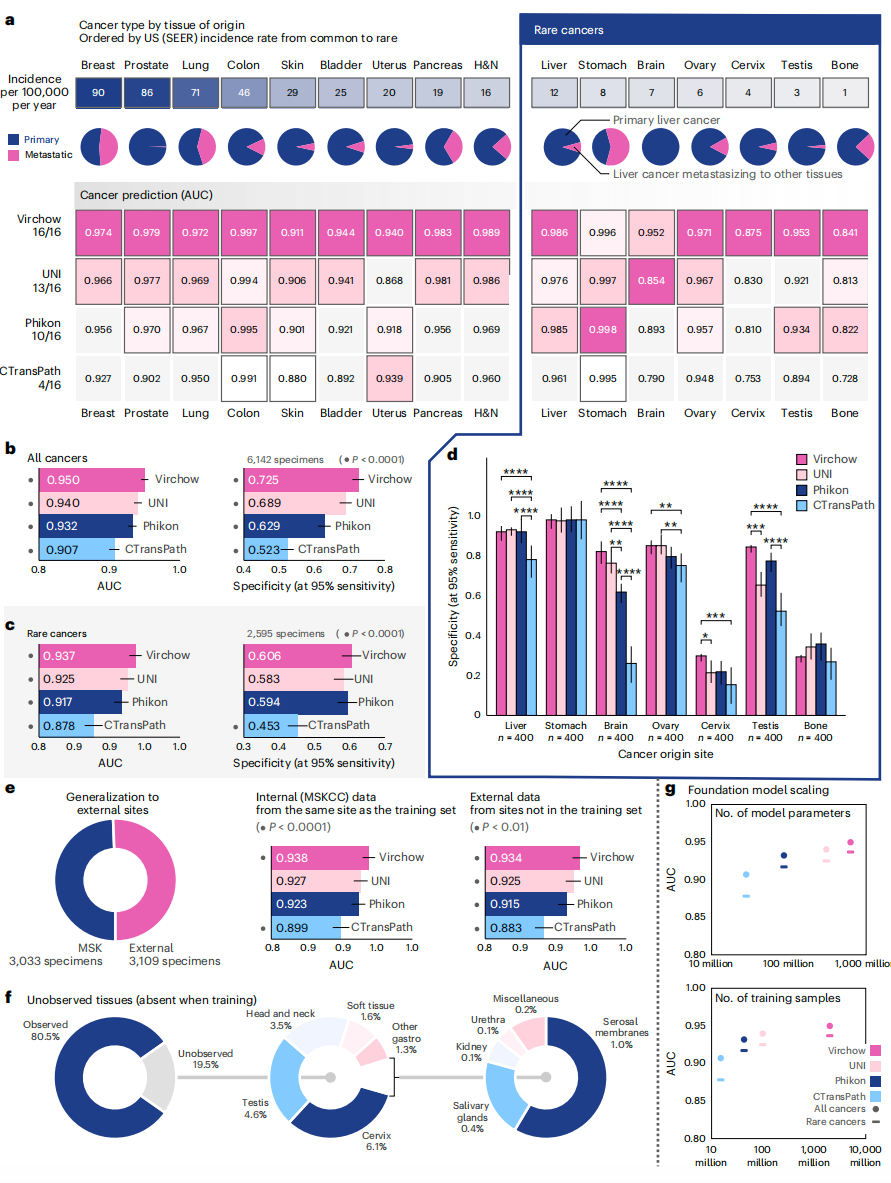
图2. Virchow模型训练泛癌检测
Virchow向临床级性能迈进
基础模型旨在提高泛化能力,但仍需要通过专业模型来验证。为此,研究团队对基于Virchow的泛癌检测模型与专业的商业模型(Paige Prostate、Paige Breast和Paige Breast Lymph Node(BLN))进行了比较,特别是前列腺癌、浸润性乳腺癌和乳腺癌淋巴结转移检测的AUC值。
虽然训练集优化不如专业模型,特定组织样本训练量也少于专业模型,但基于Virchow的泛癌检测模型表现接近专业模型,在癌症罕见变异上更优(图 3a-d)。具体来说,Paige Prostate在66,713个样本上进行训练,Paige Breast在44,588个样本上进行训练,BLN在8150个样本上进行训练,而基于Virchow的泛癌检测模型只在35,387个样本上进行训练。泛癌模型在前列腺、乳腺和BLN上的AUC分别达到0.980、0.985和0.971,接近专业模型。在检测大的转移灶方面,基于Virchow的泛癌检测模型明显优于 Paige BLN(0.999 vs 0.994 AUC,P < 0.05)。此外,在其他BLN比较或一些分层的乳腺癌比较中无统计学差异(P < 0.05)(图 3c)。
泛癌模型除了在整体AUC方面接近专业模型外,在癌症罕见变异方面表现更出色,该模型可在前列腺和淋巴结组织中检测到未训练过的淋巴瘤。在某些淋巴瘤变异和乳腺组织的罕见组织变异检测上优于专业模型,但由于相关变异罕见,这一预测的统计效力不足。
为理解泛癌模型的错误模式,病理学家检查评估WSIs中的错误案例,选择每种组织的操作点以达到高灵敏度和特异性,记录并分类错误模式,分别分析了假阳性和假阴性模式,发现假阴性病例主要由小肿瘤病灶、边界恶性潜能的肿瘤、低级别星形细胞瘤等原因导致;假阳性病例主要由前驱病变、组织伪影、间质或淋巴成分的反应性改变等原因导致。这些模式可能对类似的癌症检测研究有益,为未来基础模型和临床AI应用的改进提供了宝贵见解。
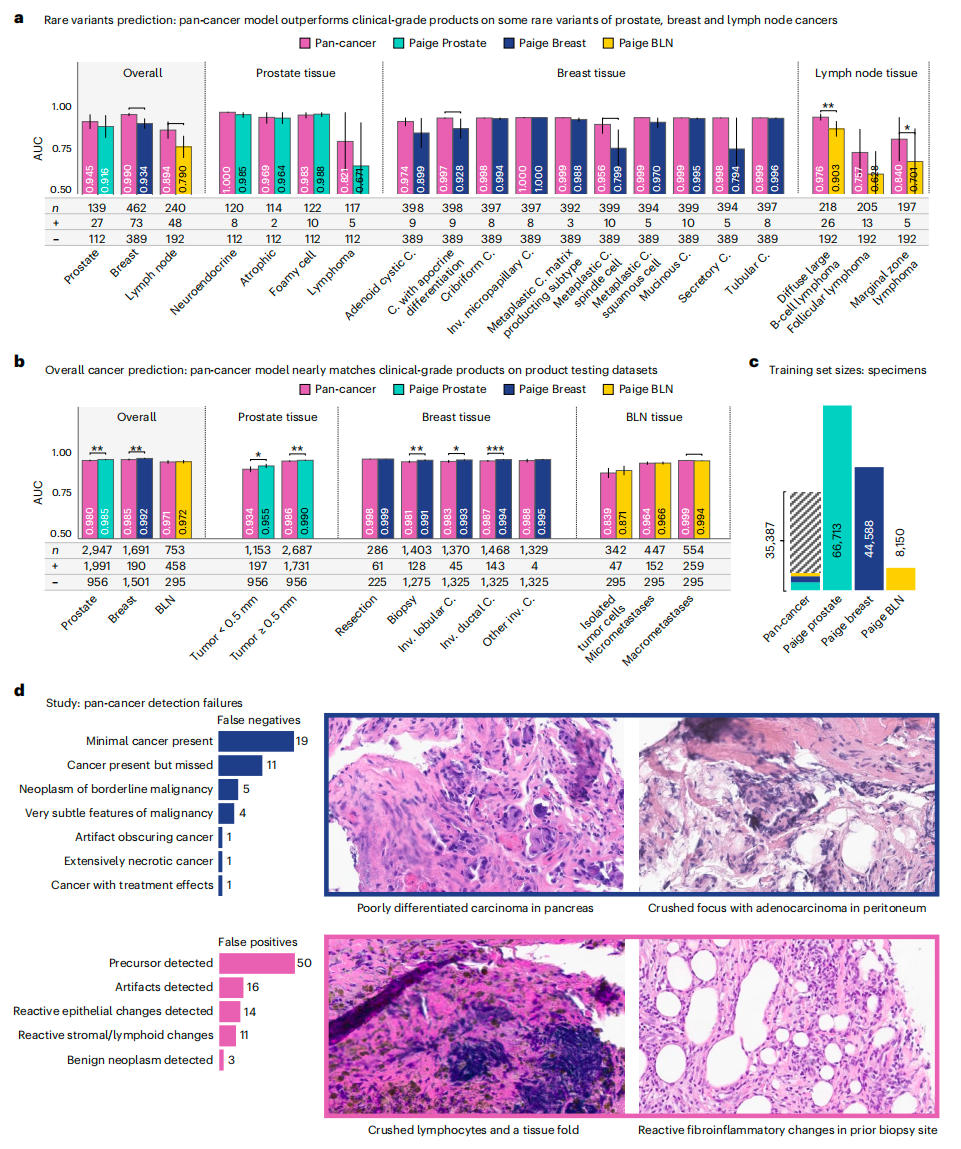
图3.泛癌检测器临床级性能检测
Virchow预测生物标志物
从标准的H&E染色图像中预测生物标记物可以减少对其他检测方法的依赖和结果返回的延迟(图 4a)。生物标记预测数据集由来自组织学切片的WSI组成,所述组织学切片与用于DNA提取和可操作靶点的MSK整合突变谱(MSK - IMPACT)测序相匹配,后者被分析以确定遗传改变的状态并建立指示变异存在或不存在的生物标志物(图 4a)。基础模型嵌入可以用来预测样本中生物标志物的状态。与泛癌评估类似,公开可用的UNI41、Phikon37和CTransPath35模型被用作比较的基线模型。
研究团队通过比较不同的生物标志物预测评估了模型性能。结果显示,在9个评估的数字生物标志物中,Virchow嵌入对其中7个的预测表现优越,AUC超越了其他基线基础模型,这强调了Virchow嵌入在多种生物标志物中的稳健性。在前列腺-雄激素受体(AR)和卵巢-基因组改变分数(FGA)类别中,Virchow嵌入的预测性能虽不是最优的,但仍是有力竞争者,AUC分别为0.849和0.847(图 4c-d)。以上结果强调了Virchow嵌入在代表H&E组织学表型方面的潜力。
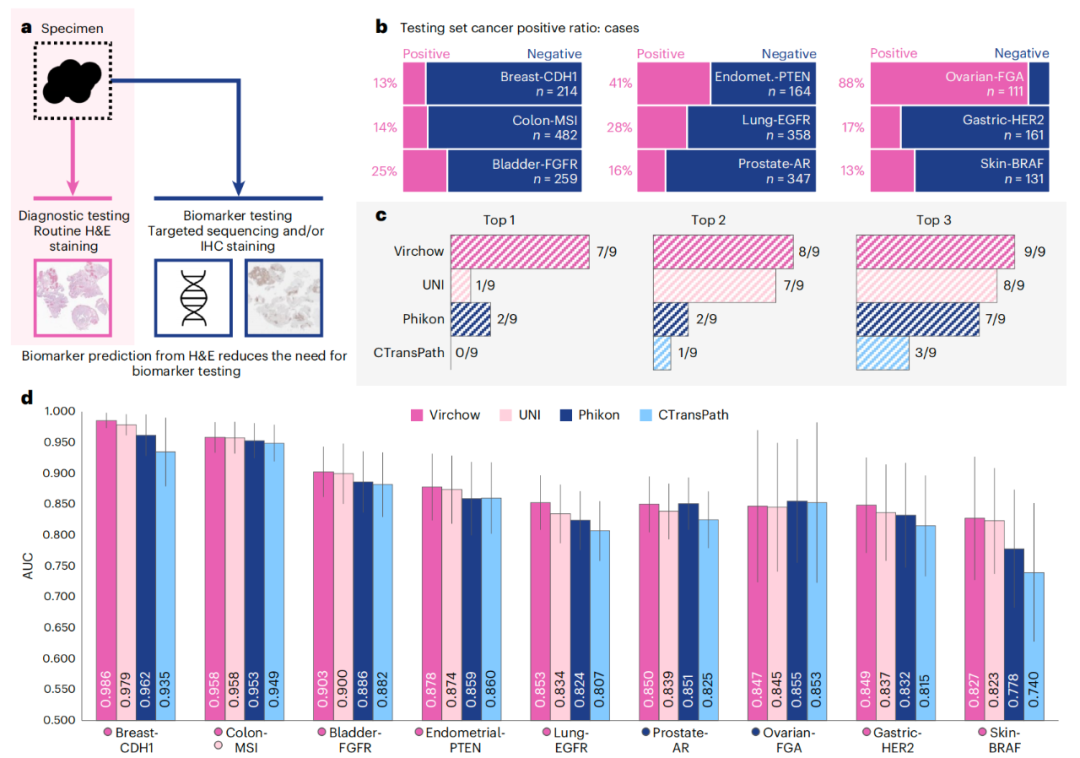
图4.生物标志物预测结果
切片级基准和定性分析
研究团队通过线性探针在一组切片级基准上评估了Virchow模型的性能,对Virchow与其他基线基础模型进行比较。该分析在公共数据集和内部MSK泛癌数据集上执行,其中内部的 PanMSK数据集是分布内基准,公共数据集是OOD基准,参与评估的基线模型除UNI、Phikon和CTransPath外,还有DINOp=8、PLIP和NatImg。结果显示,Virchow嵌入在7/8的切片级基准检测任务中表现相当或优于其他嵌入模型的性能,UNI和Phikon是其有力竞争对手(图 5a-c)。Virchow对数据预处理的变化具有鲁棒性,即使在未标准化的情况下性能仅轻微下降。此外,Virchow的无监督特征分析表明,其嵌入学习能够将图像分离成有意义的特征簇。
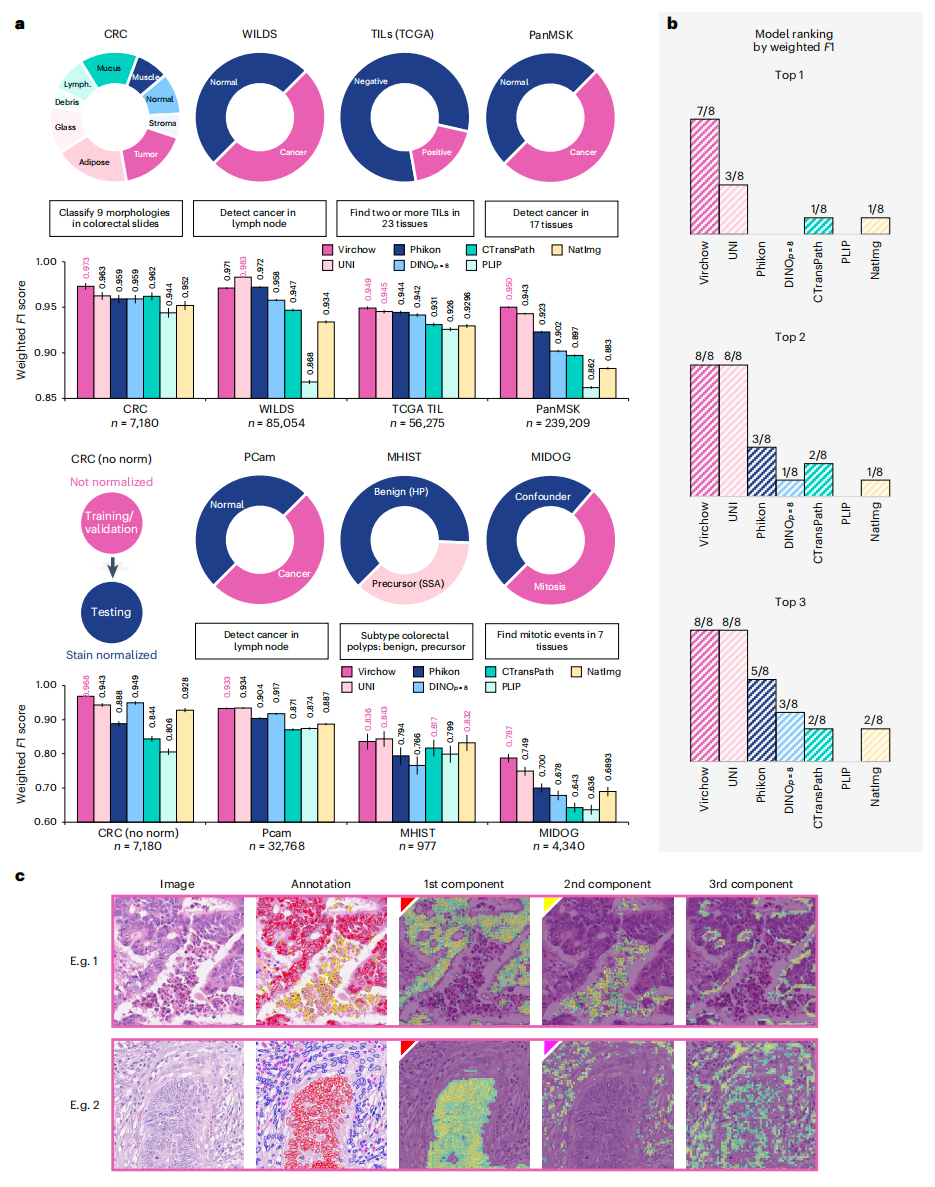
图5.切片级线性探针分析总结
综上所述,该研究展示了迄今为止计算病理学领域最大的基础模型——Virchow。Virchow模型不仅能评估生物标记物预测和细胞识别,还能实现泛癌检测,在9种常见癌症和7种罕见癌症中达到了0.95的样本级AUC。基于Virchow构建的泛癌检测即使在训练数据较少的情况下也能实现与组织特异性临床级模型相似的性能,并在一些罕见的癌症变异上表现更优。Virchow的性能提升凸显了基础模型的价值,并为标记训练数据有限的许多高影响力应用开辟了可能性。
Virchow模型可在https://huggingface.co/paige - ai/Virchow访问,用于非商业研究和复制结果的公共软件开发工具包可在https://github.com/Paige - AI/paige - ml - sdk获取。
论文原文:
Vorontsov, E., Bozkurt, A., Casson, A. et al. A foundation model for clinical-grade computational pathology and rare cancers detection. Nat Med (2024). https://doi.org/10.1038/s41591-024-03141-0


