清华大学吴励课题组《CMI》:揭示调控树突状细胞发育与生存的重要机制
时间:2024-06-04 21:03:03 热度:37.1℃ 作者:网络
清华大学基础医学院吴励教授课题组在2024年5月31日的《Cellular & Molecular Immunology》期刊上在线发表了题为“TRIM33通过对Irf8与Bcl2l11转录的调控在树突状细胞的分化与稳态中发挥重要作用”(TRIM33 plays a critical role in regulating dendritic cell differentiation and homeostasis by modulating Irf8 and Bcl2l11 transcription)的研究论文。该研究首次报告了TRIM33通过促进Irf8表达以支持cDC1发育,同时通过抑制Bcl2l11在所有DC亚群及其前体中的表达,在维持DC的稳态中起到关键作用。

作为免疫系统中重要的调控细胞,树突状细胞(dendritic cells,DC)的稳态对于维持机体的先天和适应性免疫反应至关重要[1]。本项研究在课题组既往鉴定的调控DC发育分化的关键转录因子和表观遗传修饰因子[2-5]的基础上,确定了TRIM33作为调控DC分化和生存的又一新的调节因子的重要功能。
已有研究显示TRIM33通过转录及表观调控对红细胞、淋巴细胞、单核细胞和粒细胞系的谱系生成和功能具有显著影响[6-8],但TRIM33在DC谱系中的作用仍不明确。此前研究显示TRIM33与转录因子PU.1,一个关键的DC命运调节因子之间具有相互作用[8],进一步提示TRIM33可能参与了DC的谱系调控。
为了研究TRIM33在DC分化及稳态中的作用,作者首先对Trim33fl/fl Cre-ERT2小鼠进行了表型分析,观察到诱导全身性Trim33敲除后,经典1型DC(conventional type 1 DC, cDC1)和浆细胞样DC(plasmacytoid DC, pDC)明显缺失,cDC2数量则显著减少,同时DC前体的数量和DC生成潜能也严重受损。
为进一步解释这些发现的分子基础,作者检测了几种关键转录因子的表达,发现TRIM33缺失的DC前体中Irf8,一个调控cDC1分化的关键转录因子的表达显著下降。综合CUT&Tag、co-IP/MS等分子机制研究,作者证实TRIM33通过与Irf8基因体和增强子区域结合,通过影响CDK9和Ser2磷酸化RNA聚合酶II的招募直接调控Irf8的转录。这一发现解释了DC(尤其是cDC1)分化对TRIM33依赖性。此外,作者在DC前体中鉴定到了数百个TRIM33和PU.1的共结合位点,并证实TRIM33通过与促凋亡因子Bcl2l11(Bim)的一个增强子位点结合来抑制Bcl2l11(Bim)的转录,进而抑制DC及其前体的凋亡。同时进一步分析确定了在CD11c+条件性敲除的Trim33fl/flItgax-Cre小鼠中,大多数DC亚群的细胞数显著减少,这说明TRIM33的缺失导致了终末分化DC的生存缺陷。此外,研究还显示在DC前体中同时过表达Irf8和敲低Bcl2l11可以恢复TRIM33缺失的DC前体产生cDC1的能力,但单一过表达Irf8或敲低Bcl2l11均不能产生此效果。因此,本研究鉴定了TRIM33在转录调控DC发育和维持其生存中的双重新功能,这些功能协同维持了DC的稳态。下图总结了TRIM33在DC谱系稳态中的关键作用。
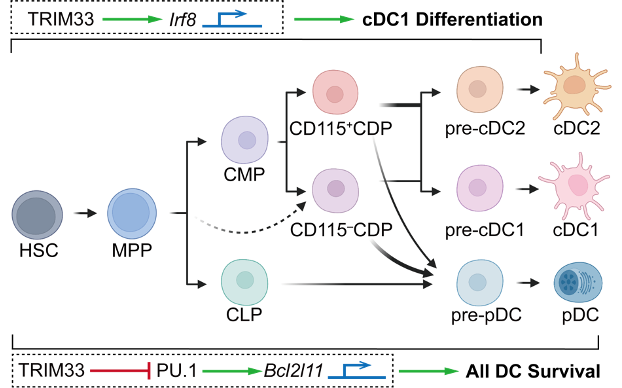
有意思的是,近期Tiniakou I. et al.发表的一篇文章也描述了非常相似的发现[9],是对本研究结果的很好佐证。该研究成果为深入了解DC发育和稳态的关键转录调节因子提供了新对知识,今后基于DC的免疫调节和治疗新策略的制定将会从这项研究中受益。
清华大学八年制医学实验班2017级学生沈相宜、基础医学院博士毕业生李晓光及博士后吴韬为共同第一作者,吴励教授为本研究的通讯作者。该研究组的已出站博士后郭婷婷、吕娇燕、何智敏、赖文龙,博士毕业生罗茂财、朱忻怡、田玉洁及清华免疫所董晨教授、胡小玉教授也为本研究作出重要贡献。本研究得到清华大学免疫学研究所实验平台、清华大学动物中心、清华大学蛋白质研究技术中心的大力支持,同时受到科技部国家重点研发计划、国家自然科学基金重大研究项目及基础科学中心,以及清华-北大生命联合中心的项目资助。
参考文献:
1. Durai, V. & Murphy, Kenneth M. Functions of Murine Dendritic Cells. Immunity 45, 719-736, doi:https://doi.org/10.1016/j.immuni.2016.10.010 (2016).
2. Wu, L., Nichogiannopoulou, A., Shortman, K. & Georgopoulos, K. Cell-autonomous defects in dendritic cell populations of Ikaros mutant mice point to a developmental relationship with the lymphoid lineage. Immunity 7, 483-492 (1997).
3. Wu, L. et al. RelB is essential for the development of myeloid-related CD8 alpha(-) dendritic cells but not of lymphoid-related CD8 alpha(+) dendritic cells. Immunity 9, 839-847, doi:10.1016/s1074-7613(00)80649-4 (1998).
4. Carotta, S. et al. The Transcription Factor PU.1 Controls Dendritic Cell Development and Flt3 Cytokine Receptor Expression in a Dose-Dependent Manner. Immunity 32, 628-641, doi:10.1016/j.immuni.2010.05.005 (2010).
5. Zhang, Y. et al. Regulation of pDC Fate Determination by Histone Deacetylase 3. eLife 12, e80477, doi:10.7554/eLife.80477 (2023).
6. Rossmann, M. P. et al. Cell-specific transcriptional control of mitochondrial metabolism by TIF1gamma drives erythropoiesis. Science 372, 716-721, doi:10.1126/science.aaz2740 (2021).
7. Wang, E. et al. The transcriptional cofactor TRIM33 prevents apoptosis in B lymphoblastic leukemia by deactivating a single enhancer. Elife 4, e06377, doi:10.7554/eLife.06377 (2015).
8. Kusy, S. et al. Adult Hematopoiesis is Regulated by TIF1 gamma, a Repressor of TAL1 and PU.1 Transcriptional Activity. Cell Stem Cell 8, 412-425, doi:10.1016/j.stem.2011.02.005 (2011).
9. Tiniakou, I. et al. Genome-wide screening identifies Trim33 as an essential regulator of dendritic cell differentiation. Science immunology 9, eadi1023, doi:10.1126/sciimmunol.adi1023 (2024).
原文链接:
https://www.nature.com/articles/s41423-024-01179-1


