Cancer Cell:基于上千例泛癌多组学数据,CPTAC发布蛋白基因组学共享数据资源
时间:2023-08-31 13:45:48 热度:37.1℃ 作者:网络
导读
全面的分子分析正在从根本上改变癌症研究。近年来,癌症基因组图谱(TCGA)和国际癌症基因组联盟(ICGC)提供了数万个肿瘤的基因组目录,为研究驱动肿瘤发生的突变奠定了基础。蛋白质是负责代谢、信号转导的主要分子,蛋白质基因组学分析是实现癌症精准治疗的有力方法,能够将基因组突变及其对细胞生理的影响联系起来。但目前领域内并未产生广泛、无偏倚的蛋白组学数据,蛋白质及其修饰数据仍严重缺乏。
临床蛋白质组肿瘤分析联盟(CPTAC)于2006年启动,是一项旨在通过应用大规模蛋白质组和基因组分析或蛋白质组学来加速了解癌症的长期研究计划。CPTAC始于美国国家癌症研究所(NCI)发起的癌症蛋白质组学技术计划(CPTC),该计划为期五年,耗资1.04亿美元,主要致力于开发和评估蛋白质组学工具和实验流程。
近日,CPTAC团队在Cancer Cell发表了题为“Proteogenomic data and resources for pan-cancer analysis”的文章,对来自10个队列的1000多个肿瘤的基因组、转录组、蛋白质组学和临床数据进行整合分析,并创建了一个强大的共享数据集用于科学分析。该文章概述了CPTAC泛癌研究团队在数据协调、数据传播和帮助生物发现的计算资源方面所做出的努力,并讨论了多组学数据整合和分析的挑战。

文章发表在Cancer Cell
主要研究内容
CPTAC主要从临床队列中生成全面的蛋白质组学和基因组学数据。目前,CPTAC数据集包括10个前瞻性肿瘤队列,包含基因组学、转录组学、蛋白质组学和磷酸化蛋白质组学分析,并提供了标准的临床/人口统计数据和组织学图像,例如性别、年龄、肿瘤分级及分期等。此外,为实现泛癌综合分析,保持一致性和可重复性,研究团队创建了一个数据集框架,对所有蛋白质基因组数据进行了重新处理。
CPTAC数据集是癌症研究的重要资源,基于该数据集进行深度科学探究和数据再分析是研究的一个初衷。为促进数据共享,CPTAC团队还提出了数据协调的计算方法和多种传播机制,以共享原始及处理后的数据。
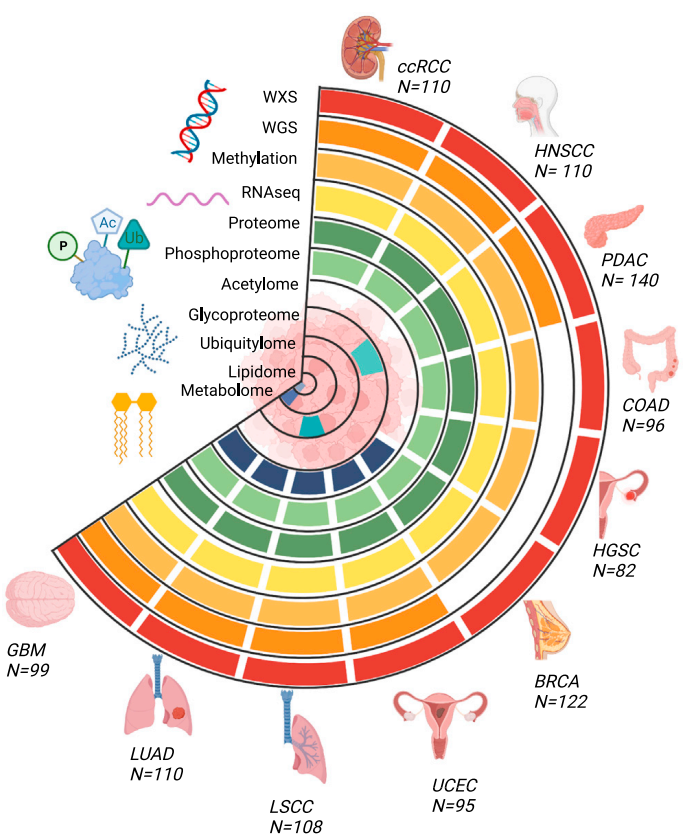
图1. CPTAC泛癌数据集的肿瘤类型和数据类型。
数据共享和集成分析
CPTAC计划生成的蛋白质基因组数据通过基因组数据共享(GDC)和蛋白质组学数据共享(PDC)进行公开传播。作为NCI云资源的组成部分,GDC、PDC与其他NCI研究数据共享资源完全集成,促进基于云的蛋白质组、基因组和成像数据分析。PDC主要由CPTAC项目驱动,通过强大的数据模型来组织数据,保持数据和相关元数据的一致性和完整性,并提供了一个界面以过滤、查询、搜索和可视化蛋白质基因组数据。
除主题数据库外,NCI的癌症研究数据共享还包含一个与数据类型无关的资源,即癌症数据服务(CDS)。存储在CDS中的CPTAC数据包括用于泛癌症分析的所有统一的蛋白质基因组数据,以及衍生分子数据。
CPTAC由具有不同数据类型专长的多个小组组成,对同一组数据应用不同的软件工具可能会导致不同的结果。因此,基准测试对于工具的评估和选择非常重要。为比较蛋白质组学数据量化的不同方法,研究团队开发了OmicsEV,通过十多个评价指标来全面评估数据深度、数据规范化、批处理效应、生物信号、平台重现性和多组学一致性。
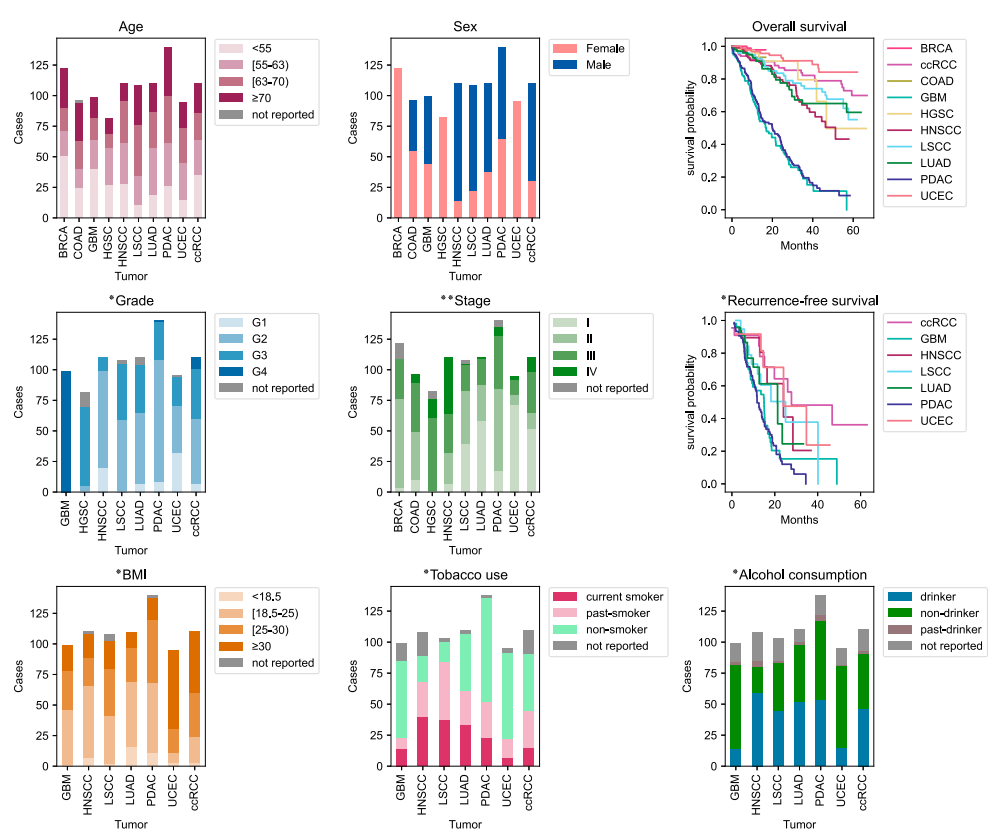
图2. CPTAC泛癌数据集的展示。
程序数据存取
简化数据访问可以显著消除使用障碍,并提高数据透明度和可重现性。CPTAC团队开发了一个软件包,将最终的定量数据表作为数据帧变量直接融入到编程环境中,可与SciKit-learn、PyTorch等常见的机器学习和可视化包集成。
此外,研究团队还将CPTAC数据集连接到其他大型公共数据集中,以扩展其效用。除利用Bioconductor中众多可用的软件工具外,用户通过TCGAbiolinks工具也可以轻松访问TCGA、GENIE和MET500等资源的分子数据。
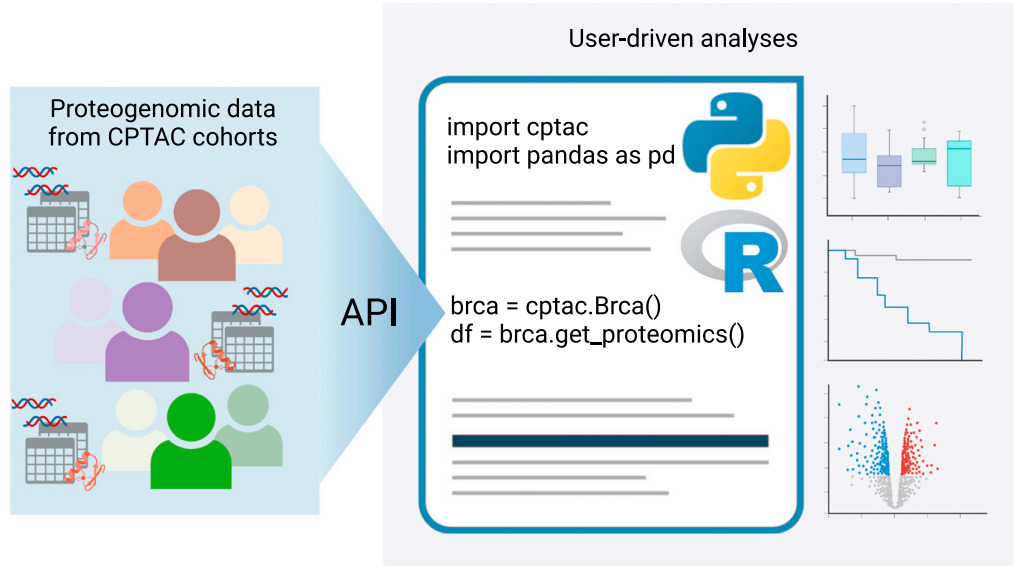
图3. 程序化访问CPTAC蛋白质基因组学。
用于数据可视化和分析的Web门户
CPTAC团队创建了多个门户网站,用于可视化及探索泛癌蛋白基因组数据。其中,每个网站均可从数据概要中提取适当的数据集,用于泛癌分析。
1 PepQuery
PepQuery(http://www.pepquery.org)允许使用MS/MS数据对基因组突变进行快速和简单的蛋白质组学验证。近期,研究团队还引入了一种新的数据索引算法来提高搜索速度,并扩展了PepQuery Web服务器的数据集。在该网站,用户可用感兴趣的新肽或DNA序列直接查询CPTAC和其他MS/MS数据,以寻找支持的肽谱匹配(PSM)。
2 LinkedOmics和LinkedOmicsKB
LinkedOmics(http://www.linkedomics.org)是一个数据分析门户网站,允许使用来自TCGA和CPTAC的癌症多组学数据表征任何目标临床或分子特征。对于每一项CPTAC研究,数据库存储了超50万个属性的数据。使用三个分析模块可挖掘上述数据,以揭示遗传突变的结果,表征基因和PTM功能。
LinkedOmicsKB(https://kb.linkedomics.org)是基于LinkedOmics开发的新知识门户网站,通过一次查询即可获得单个基因和表型的预先计算结果,具有用户友好的可视化功能,便于理解。
3 PTMcosmos
PTMcosmos(https://ptmcosmos.wustl.edu/)是一个交互式的Web门户网站,旨在对人类PTM进行分类和可视化。该网站允许人们能够查询现有PTM位点相关文献、肿瘤和正常样本之间的丰度差异以及蛋白质结构上的PTM突变簇。
4 ProTrackPath
ProTrackPath(http://pancan.cptac-data-view.org/)可用于访问泛癌队列中的通路富集评分。ProTrackPath可通过单样本基因集富集分析(ssGSEA)计算不同癌症类型的通路富集评分;用户指定一个通路数据库并选择一组通路进行可视化,最终可生成一个交互式热图。
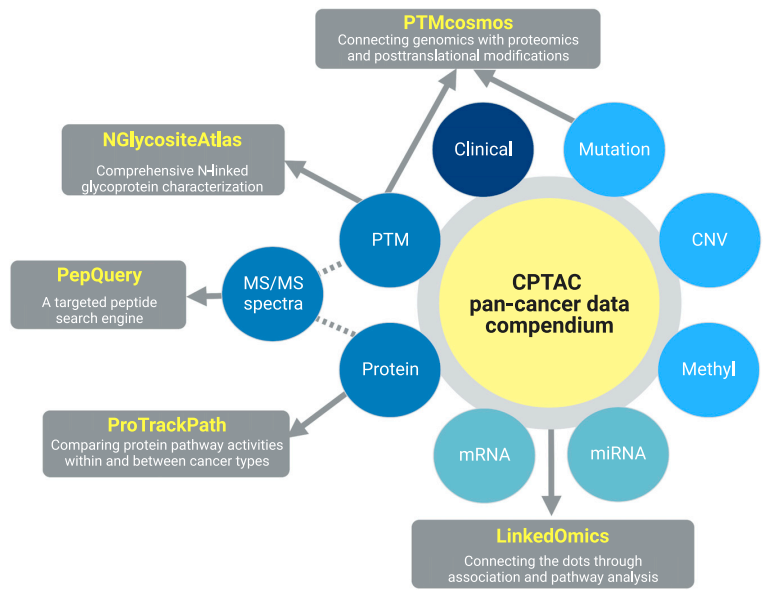
图4. 用于数据可视化和分析的Web门户。
结 语
综上所述,研究团队重新处理了来自CPTAC的10个癌症队列的数据,创建了一个泛癌蛋白基因组数据集;并介绍了用于创建该数据概要的方法、数据访问的方法以及泛癌症多组学数据分析的关键等。目前,CPTAC数据库资源已公开共享,研究团队希望其能推进癌症诊断和治疗。
参考文献:
Li Y, Dou Y, et.al,. Proteogenomic data and resources for pan-cancer analysis. Cancer Cell. 2023 Aug 14;41(8):1397-1406.
https://www.cell.com/cancer-cell/fulltext/S1535-6108(23)00219-2


