PNAS:基于甲基化图谱及深度学习模型,实现cfDNA组织特异性定性及定量研究
时间:2023-07-25 11:21:16 热度:37.1℃ 作者:网络
组织中死亡的细胞会将其DNA作为细胞游离DNA(cfDNA)释放到血液中。因此,解析cfDNA的组织起源,在辅助疾病诊断、预后和治疗监测方面具有巨大的临床潜力。cfDNA检测提供了一种非侵入性的、全面的身体所有组织的健康状况检测的方法。
由于DNA甲基化的组织特异性,cfDNA可以根据其甲基化模式追溯到其起源的组织,一些研究提出了基于甲基化的组织反褶积方法来估计组织特异性cfDNA的比例。虽然cfDNA的反褶积研究前景广阔,但仍面临着挑战:1) 来自实体器官的cfDNA仅占cfDNA的一小部分,因此cfDNA中病理器官的信号通常较弱;2) 体内所有组织都可以释放cfDNA,为了得到准确的组织反褶积,需要尽可能多组织类型的联合反褶积,而不是只对少数组织类型进行联合反褶积。
近日,加州大学洛杉矶分校研究团队与斯坦福大学团队、EarlyDiagnostics Inc公司团队等合作在PNAS发表了题为“Comprehensive tissue deconvolution of cell-free DNA by deep learning for disease diagnosis and monitoring”的文章。为了充分利用组织cfDNA的临床潜力,研究团队报告了一个基于521个非癌症组织样本的全面的高分辨率组织甲基化图谱,涵盖29种主要的人体组织类型。基于丰富的组织甲基化图谱,研究团队开发了第一个cfDNA监督组织反卷积方法,一个基于深度神经网络(DNN) 的模型cfSort,其可用于敏感和稳健地定量cfDNA中的组织成分。该甲基化图谱解决了组织特异性甲基化研究的缺口,cfSort模型解决了现有反褶积方法的技术局限性,有助于提高cfDNA组织反褶积的敏感性和准确性,提高组织衍生cfDNA的临床应用价值,将在疾病检测和监测中具有广泛的研究和临床应用。

文章发表在PNAS
主要研究内容
01 构建全面的组织甲基化图谱
研究人员纳入了来自基因型-组织表达(GTEx)项目的521个非癌症个体组织样本的甲基化数据(RRBS),涵盖了29种主要的人体组织。基于这些RRBS数据,研究人员在DNA片段水平上系统地发现了组织特异性甲基化标记物。
为了构建一个全面的组织甲基化图谱,研究人员采用了三种策略,得到了三种可以覆盖几乎所有差异组织模式的标记类型。对于每种策略,研究人员对标记物在组织样本中的一致性进行排序,即显示组织特定甲基化模式的样本数量。三种策略得到的组织特异性甲基化标记物相互补充。最后将这些标记物结合起来构建组织特异性甲基化图谱。

图1. 筛选组织特异性甲基化的三种策略。
02 甲基化图谱的组织特异性验证
研究团队从四个方面用独立的数据源验证了组织标记图谱的准确性。1)组织特异性甲基化的可重复性。组织标记物中有92.9%在表观基因组项目的全基因组亚硫酸氢盐测序 (WGBS) 数据中显示了一致的组织特异性甲基化,表明组织甲基化图谱捕获了真实的组织特异性甲基化模式;2)与组织特异性组蛋白修饰的关联。研究人员关注的是H3K27ac修饰,观察到93.7%的组织标记物有一致的组织特异性H3K27ac修饰;3)与组织特异性基因表达的关联。在GTEx项目的转录组测序数据中,当相应的启动子区域在组织类型中被低甲基化时,63.0%的组织标记物增加了基因转录水平,意味着组织特异性甲基化可能影响组织特异性基因表达;4)与组织特异性转录调控的关联。研究人员对转录进行了富集分析并在组织标记物上发现了转录因子结合基序,这些基序主要与发育、分化和组织特异性表达有关。因此,通过使用独立数据集的这四项验证,表明了该图谱中的组织甲基化标记物具有组织特异性和生物学意义。
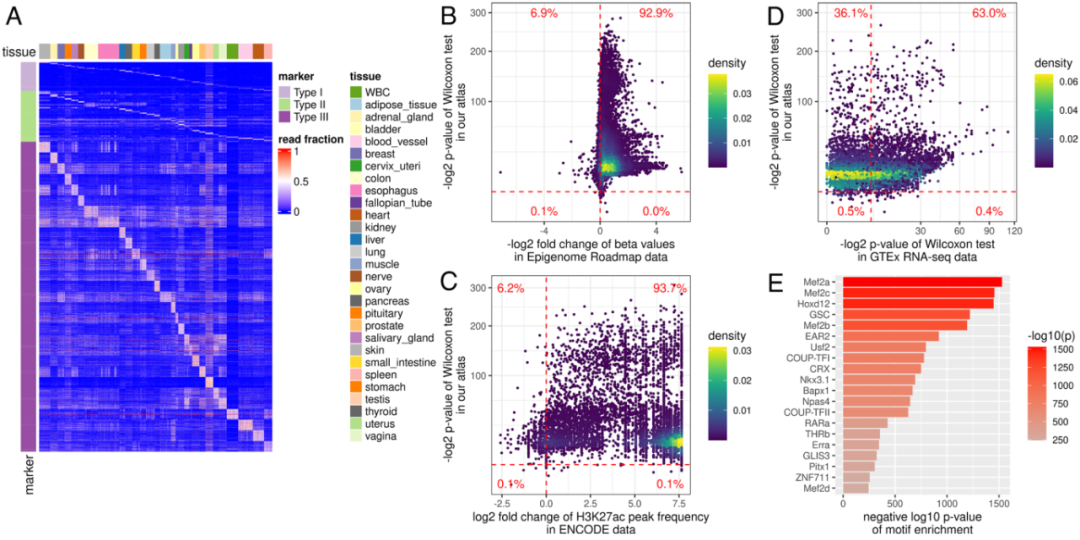
图2. 组织特异性甲基化图谱的构建和验证。
03 cfSort模型概述
研究人员使用521个组织样本的RRBS数据,以预定义的组织组成和不同覆盖深度生成cfDNA甲基化数据集作为训练、验证和测试数据。研究共生成295484个不同的训练样本和数千个验证和测试样本,满足DNN训练和评估的要求。此外,在数据生成中,研究人员充分利用不同组织类型、不同样本、不同组织组成的组合,探索数据中可能存在的噪声和偏差。因此,cfSort将从这些数据中学习稳健的组织特异性。
cfSort的基本结构是两个DNN的集合。每个DNN以特征集合作为输入,输出29种组织类型的预测组织组成。对于每个DNN,研究人员构造了三个节点数减少的密集隐藏层,并使用整流线性单元作为激活函数。隐藏节点可以自动学习权重,对输入特征进行优先排序,使DNN能够抵抗数据中的噪声。考虑到训练数据的大小和多样性,研究人员在每个密集层之前增加了一个批处理归一层,以稳定和加速训练过程。此外,还对DNN进行正则化,以增加模型的鲁棒性,避免过拟合。最终将预测的组织组成计算为两个DNN的平均预测。
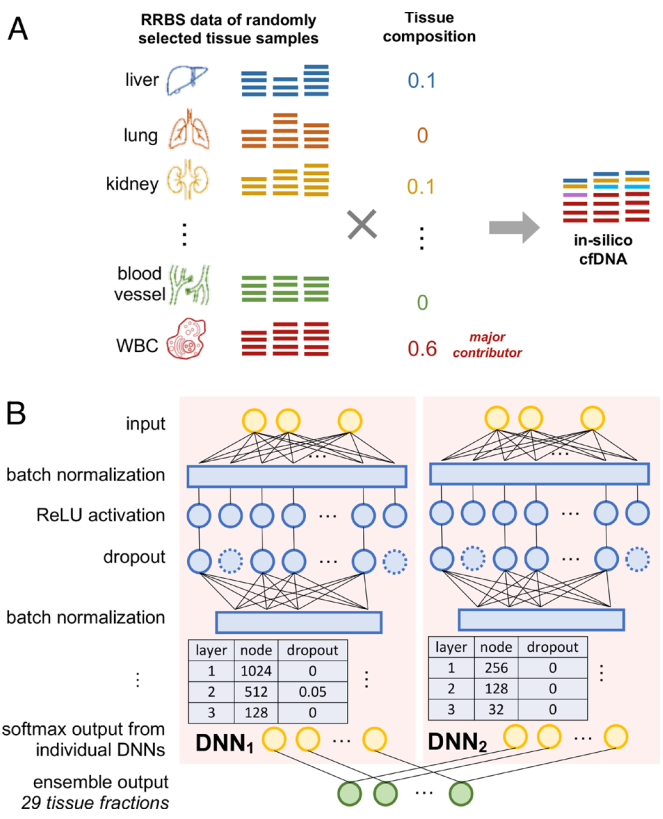
图3. cfSort分析流程图。
04 cfSort的分析性能
研究团队在一个独立的测试集上评估了cfSort的性能,将其性能与两种现有的组织反褶积方法进行了比较:非负最小二乘法(NNLS)和CelFiE。使用平均绝对误差、估计的组织分数与真相之间的相关性来评估方法的准确性。cfSort在所有指标上都优于NNLS和CelFiE,表明cfSort在估算组织成分方面的准确性高于其他两种方法。
反褶积方法需要有较高的检测限,以低比例检测组织衍生的cfDNA。为了评估cfSort的检测限,研究团队采用了一种广泛使用的系列稀释方法。对于系列稀释中的每个样本,将单个组织样本与检测组中已知组织分数和不同覆盖深度的样本混合。cfSort能够以0.1%的组织分数检测组织衍生的cfDNA,而NNLS以5%,CelFiE以0.5%检测组织衍生的cfDNA。以上结果表明cfSort比两种竞争方法具有更好的检测下限。
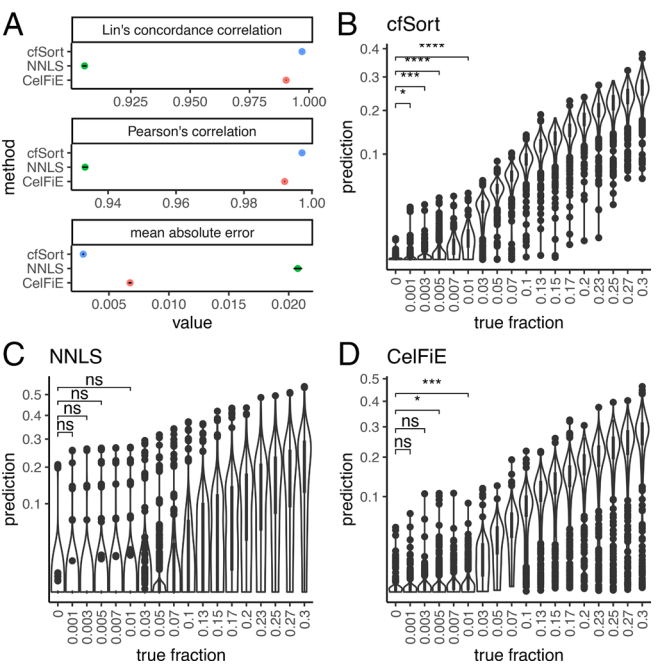
图4. cfSort分析性能的评估与比较。
05 cfSort在临床中的应用
解析cfDNA中的组织成分可以揭示受影响组织内稳态的改变。研究人员将cfSort应用于健康和患病个体的cfDNA甲基组数据,以分析cfDNA中的组织组分是否可以指示疾病的发生率。
在所有的比较分析中,发现患者的受影响组织分数显著高于健康个体。此外,受影响的组织分数随着癌症分期的增加而增加。研究人员还使用受影响的组织分数作为唯一的预测因子来评估疾病,结果显示,cfSort可以检测来自病变组织的升高cfDNA,表明cfSort在辅助疾病诊断和监测治疗方面具有广泛的临床应用价值。随后,利用肝癌和健康人cfDNA WGBS数据进一步验证了cfSort对不同平台甲基化数据的适用性,揭示疾病引起的组织成分变化。
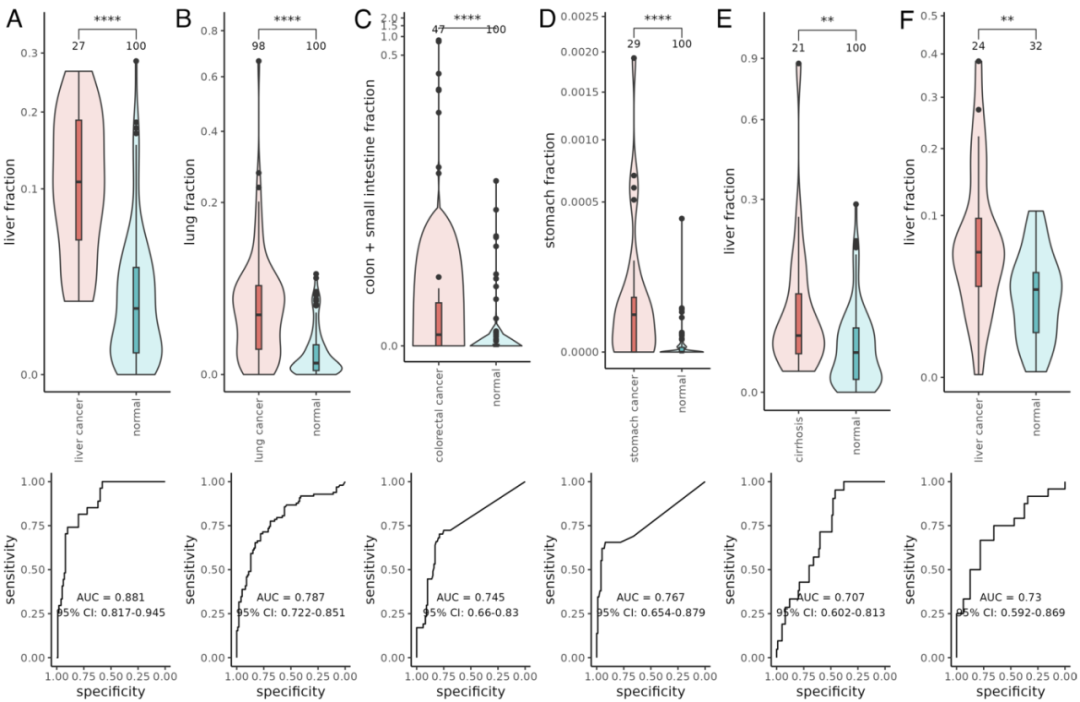
图5. cfSort用于分析患者和健康个体的cfDNA。
结 语
综上所述,该研究报告了一个全面的高分辨率组织甲基化图谱和基于cfDNA的首个监督型组织反卷积方法—cfSort,其能够对cfDNA中的组织部分进行敏感而稳健的定量。研究人员还通过多个独立的数据集验证了组织标记图谱和cfSort的性能,表明这些标记与组织发育、组织分化和组织特异性转录相关。组织甲基化图谱和cfSort增强了cfDNA中组织去卷积的性能,从而促进了基于cfDNA的疾病检测和纵向治疗监测。
文章第一作者、加州大学洛杉矶分校Shuo Li表示:“cfSort是一种监督计算方法,可以确定cfDNA样本的组织来源。监督式机器学习依赖于输入和输出训练数据,而非监督式学习则依赖于原始数据。有了监督学习,模型可以吸收尽可能多的训练数据,训练数据越多,预测就越准确。”
据悉,cfSort已经申请了一项临时专利,该团队目前正在计划将该方法应用于不同疾病类型的更多研究。
参考资料:
Li S, et al., Comprehensive tissue deconvolution of cell-free DNA by deep learning for disease diagnosis and monitoring. Proc Natl Acad Sci U S A. 2023 Jul 11;120(28):e2305236120.

