Genome Med综述 | 长读长测序技术在孟德尔遗传学中的应用及展望
时间:2023-07-05 13:06:24 热度:37.1℃ 作者:网络
导读
近年来,随着基因检测技术的高速发展,尤其是外显子组测序在临床实践中得到广泛应用,许多罕见遗传疾病的分子病因已被揭示,但仍有约一半的遗传疾病患者(孟德尔遗传病)未得到精确的分子诊断。短读长测序(SRS)准确性较高、覆盖率更均匀,但不能准确地将reads映射到基因组的重复区域。已有研究表明,许多较大、复杂的人类遗传变异形式(例如结构变异SV)会被SRS和外显子组测序遗漏。
长读长测序(LRS)可用于评估遗传病因不明或具有复杂变化的个体,既能提高诊断率,又能缩短精确遗传诊断所需的时间,是一种颇具前景的技术。近期研究表明,与SRS相比,LRS可以更准确地检测250多种医学相关基因,并能解决复杂的SV、重复扩增以及甲基化差异。这提示将LRS系统地应用于先前未解决的孟德尔病例可能会提高总体诊断率。
近日,来自美国华盛顿大学霍华德休斯医学院的研究团队在Genome Medicine上发表了题为“Applications of long-read sequencing to Mendelian genetics”的综述文章。该文章总结了当前的LRS技术,概述了其在临床研究和人类健康方面的实用性和优势,并讨论了LRS未来的临床应用。随着成本的不断降低,LRS将在临床领域发挥重要作用,将从根本上改变病理变异的发现方式,最终为临床提供服务。

文章发表于Genome Medicine
目前,市场上的LRS平台主要由Pacific Biosciences(PacBio)和Oxford Nanopore Technologies(ONT)生产,均可产生大于10kbp的RNA或DNA reads,两种技术的序列数据生成方式存在根本差异,进而导致了输出率和错误率的差异。(图1)。与SRS相比,PacBio提供了更高的测序准确性,ONT提供了更长的reads(>2Mbp)、快速周转和直接RNA测序。
PacBio可提供两种测序模式:最初为最大限度地增加序列reads长度开发了continuous long-read(CLR)测序,通常涉及制备长度大于30kbp的文库;2019年为提高准确性推出了Circular Consensus Sequencing(CCS)模式,使用高保真(HiFi)reads。
PacBio目前专注于CCS测序模式,其靶向较短的DNA片段(10 - 30kbp),并在DNA片段两端连接一个hairpin adapter(称为SMRTbell),从而形成环状分子;DNA聚合酶在分子的反向链和正向链上进行多次迭代产生高精度HiFi reads。随着CCS的引入,PacBio测序的准确性已与Illumina相当,大多数残留误差仅限于均聚物中的小片段插入或缺失(InDel)。
ONT测序是新一代基于纳米孔的单分子实时电信号测序技术,其工作原理是DNA/RNA链在马达蛋白的带领下与镶嵌在生物膜上的纳米孔蛋白结合并解旋,在生物膜两侧电压差的作用下,DNA/RNA链以一定的速率通过纳米孔通道蛋白,引起不同电信号的变化;通过对这些信号进行检测及对应,即可获得相应碱基的类型,完成序列的实时测定。此前,与SRS或PacBio HiFi测序相比,ONT测序的准确性较低。近年来随着化学、孔隙设计和碱基调用模型的改进,ONT测序单核苷酸变异(SNV)的30倍覆盖率为99.4%,InDel召回率为63-68%。
ONT测序有几个独特优势,一是具有自适应采样的测序模式,可在测序过程中实现基因组特定区域的富集或缺失。与PacBio一样,ONT平台支持互补DNA(cDNA)测序,其另一个独特的方面是能够使用专用试剂盒直接测序天然mRNA分子,这允许直接测量poly-A尾部的长度,检测mRNA修饰。
除上述多种技术外,目前也已开发了几种长读长测序纠错工具,可以分为混合工具(Hercules、proovread等)和基于自校正的工具(FLAS和LoRMA)。经比较分析,混合工具优于非混合工具,尤其是在LRS覆盖率较低的情况下。

图1. PacBio和ONT的文库制备和测序工作流程
使用LRS识别变异最直接的方法是基于read(read-based ),即依据参考基因组对reads进行映射,由于reads长度通常比大多数常见的重复序列长(>10kbp),该方法显著提高了SV检测的灵敏度。随着专门的比对工具(BLASR、MHAP和Minimap2等)以及专门用于变异发现和定相的软件(WhatsHap、DeepVariant和Sniffles等)的发布,使用LRS的基于reads的映射方法得到了改进、优化,能够处理更长、更容易出错的数据。
原则上,基于长读长数据集的基因组从头组装可以确定一个样本两种单倍型的完整或接近完整的端粒-端粒(T2T)DNA序列,基于此,科研人员近期已开发了几种基因组组装器。基因组组装提供了人类基因组的最完整表征,并具有研究人类遗传变异全图谱的潜力,从SNV到全序列解析的SV,包括拷贝数变异(图2)。
2021年,人类基因组结构变异联盟(HGSVC)利用CLR、HiFi PacBio和Strand seq技术,成功组装了32个人类基因组样本(64个单倍型)的单倍型参考基因组。该研究团队开发了一种定相组装变异(PAV)调用程序,首次通过将单个样本的两个单倍型与人类参考基因组进行直接比较来发现变异(SNV、InDel、SV)。该研究在普通人群中发现了超过100,000个SV,并提供了第一个全面的人类基因组结构变异与侧翼SNV连锁不平衡的序列解析图谱,促进了新的表达数量性状位点和疾病关联的发现。2022年,人类泛基因组参考联盟(HPRC)使用HiFi PacBio和亲子Illumina WGS数据,从47个人类基因组(94个单倍型)中组装了一个更完整的泛基因组。虽尚未完成,但HGSVC和HPRC绘制的SV目录,有助于发现人类疾病样本中的潜在致病变异。
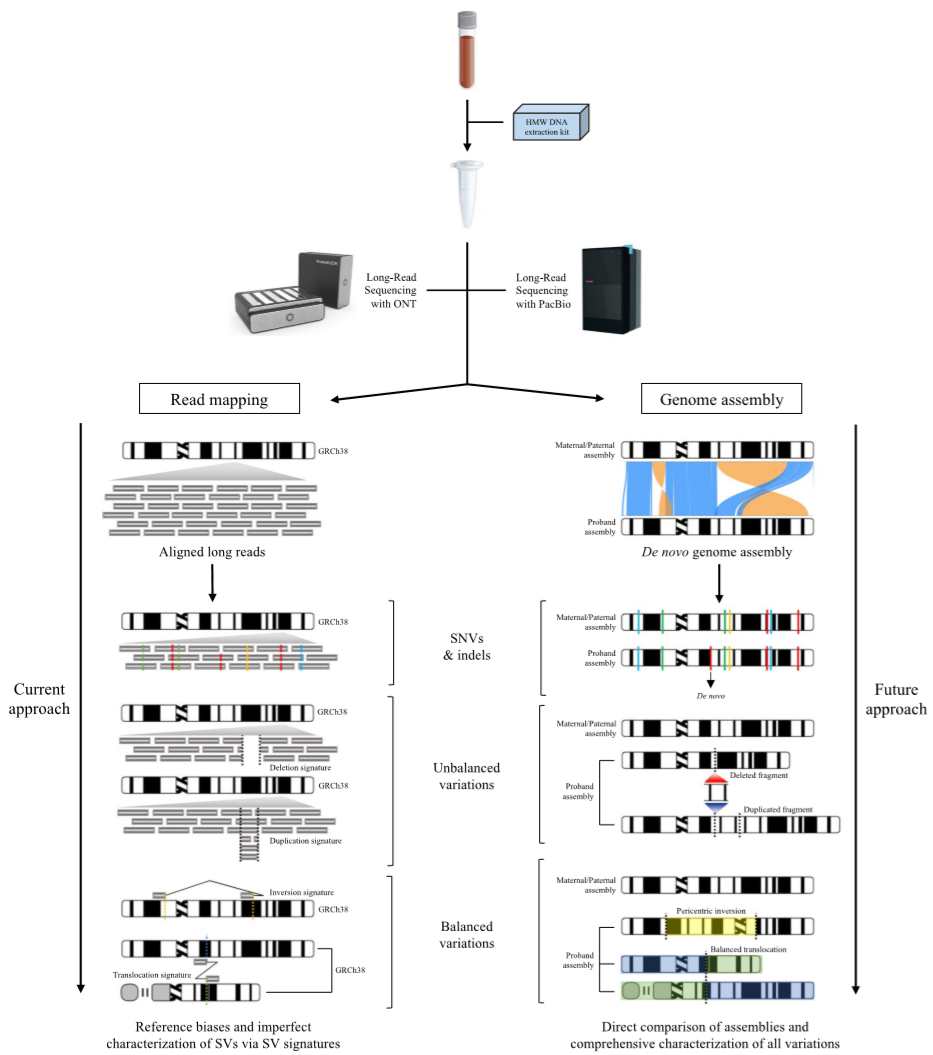
图2. 变异识别与基因组组装
除发现遗传变异外,LRS还提供了与更广泛健康相关的新的生物学见解。ONT测序具有对cDNA和天然RNA分子进行测序的潜力,已被用于研究细菌和病毒的RNA以及人类的RNA。PacBio可用于cDNA测序,其全长异构体测序方案称为Iso-Seq,已成功用于表征剪接事件、检测融合基因,并在整体和单细胞/单核水平上鉴定组织和等位基因特异性异构体。
在过去几年中,使用PacBio或ONT成功识别临床检测遗漏的致病性变异的报告数量急剧增加,其中LRS最大的优势在于识别与疾病相关的SV。这提示LRS的应用将有助于研究未解决的孟德尔病例,特别是外显子组测序和SR-WGS阴性的病例。
研究团队使用PacBio测序技术评估了六名患有神经发育障碍的患者及其健康父母(图3)。LRS分析显示,患者1的CDKL5中出现了7kbp的新插入;患者2出现了复杂的新SV;患者4中发现了一个影响6、7和9号染色体的大型复杂的新发SV。这些结果表明,科研人员能够通过LRS深入研究孟德尔病例,改善患者预后;长读长WGS(LR WGS)有望作为单一临床试验成为SR WGS的潜在替代方案。
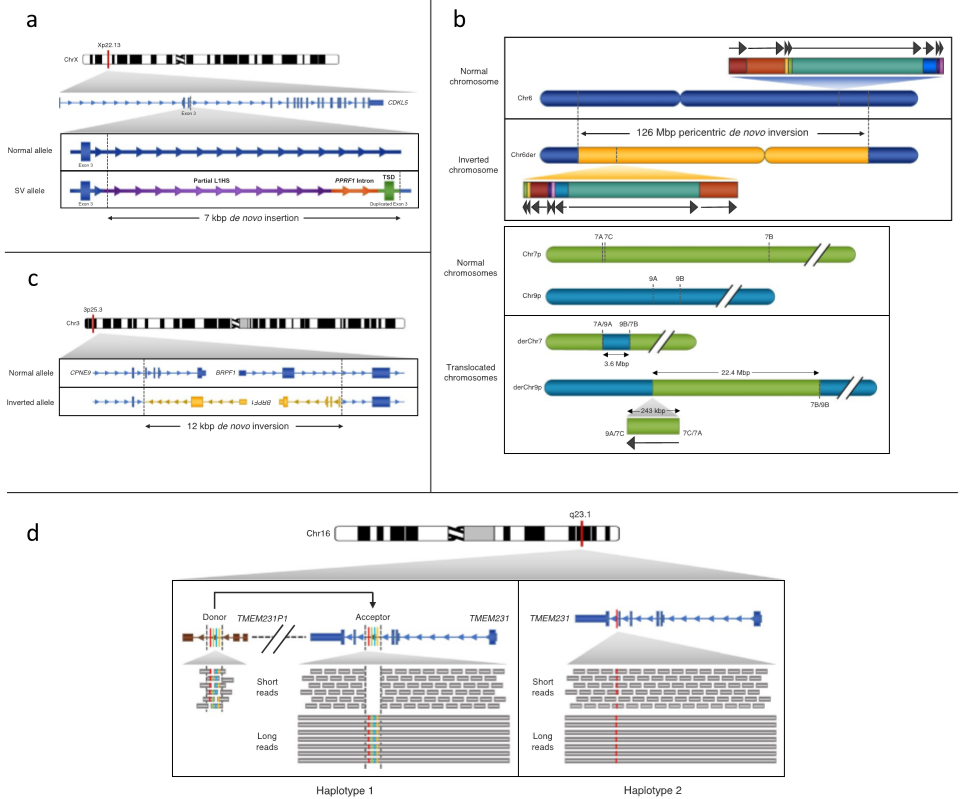
图3. 以LRS为特征的SNV和SV的示例
综上所述,该文章总结了当前的LRS技术,概述了其如何被用于评估复杂的遗传变异并识别缺失的变异,并讨论了LRS在近期和长期内如何在临床环境中使用。LRS改进了变异发现(尤其是SV)、基因组物理定相、在没有额外实验的情况下同时发现甲基化差异和遗传变异,以及基于临床怀疑重新分析单个数据集的能力。此外,LRS还揭示了可能适用于定向治疗的新生物过程,并允许开发改进的临床测试,以减少进行精确基因诊断所需的时间。
参考文献:
Mastrorosa FK, Miller DE, Eichler EE. Applications of long-read sequencing to Mendelian genetics. Genome Med. 2023;15(1):42. 2023 Jun 14. doi:10.1186/s13073-023-01194-3


