Genome Biol:突破传统直接RNA测序局限!新方法可同时检测多类ncRNA和mRNA
时间:2024-09-20 19:02:15 热度:37.1℃ 作者:网络
RNA测序技术自问世以来经历了多次迭代,但这些技术在RNA修饰鉴定、RNA结构和剪接变化分析方面仍存在挑战。近年来,PacBio和Oxford Nanopore Technologies(ONT)等公司的测序平台不断发展,尤其是Nanopore平台可直接对自然RNA链进行测序,并简化文库制备和分析过程。
目前,科研人员已表征超100种独特的RNA修饰,包括腺苷到肌苷(A-to-I)编辑、假尿嘧啶修饰等;其中,非编码RNA(ncRNAs)是这些修饰的常见靶点,对人类健康和疾病具有重要意义。Nanopore平台的直接RNA-seq方法在RNA修饰检测分析方面具有潜力,但其在文库制备过程中使用poly(A)选择的标准直接RNA-seq方法,这限制了对许多不同类型ncRNA的捕获。
近日,加拿大曼尼托巴大学的科研团队在Genome Biology发表了题为”NERD-seq: a novel approach of Nanopore direct RNA sequencing that expands representation of non-coding RNAs”的文章。研究团队提出了一种名为NERD-seq的方法,扩展了标准直接RNA-seq的应用范围,能够在全基因组范围内检测多种ncRNA类型,同时保持了对高复杂性mRNA转录组的测序能力。使用神经组织作为验证,该研究证明NERD-seq技术扩展了频繁修饰ncRNA的表征,包括snoRNA、snRNA、scRNA、srpRNA、tRNA和rRF等。综上,NERD-seq能够同时研究脑组织及其他组织中的mRNA和ncRNA表观转录组。

02 NERD-seq方法
标准的直接RNA-seq方法依赖于poly(A)选择,可对poly(A)转录组(主要是mRNA)进行测序,但无法有效检测没有poly(A)尾的短ncRNA。为改善这一局限性,同时保持对poly(A)转录组进行测序的能力,研究团队开发了一种名为NERD-seq的非编码富集RNA直接测序方法(图1)。
该方法利用柱基大小富集将总RNA分为长RNA和短RNA两部分,短RNA部分通过poly(A)聚合酶添加poly(A)尾部,使其能够与poly(T)测序接头蛋白形成碱基配对,从而提高短RNA纳米孔测序的准确性;长RNA部分则保留全长rRNA,避免多聚腺苷酸化;在灭活poly(A)聚合酶后,将两个部分(短和长RNA)再次聚集在一起。
此外,为克服高度结构化RNA区域在测序过程中的阻碍,研究团队使用GspSSD 2.0 DNA聚合酶改进了互补链的合成。经验证,NERD-seq能够同时富集短ncRNA、mRNA和长ncRNA,而不会产生显著的poly(A)长度偏差。
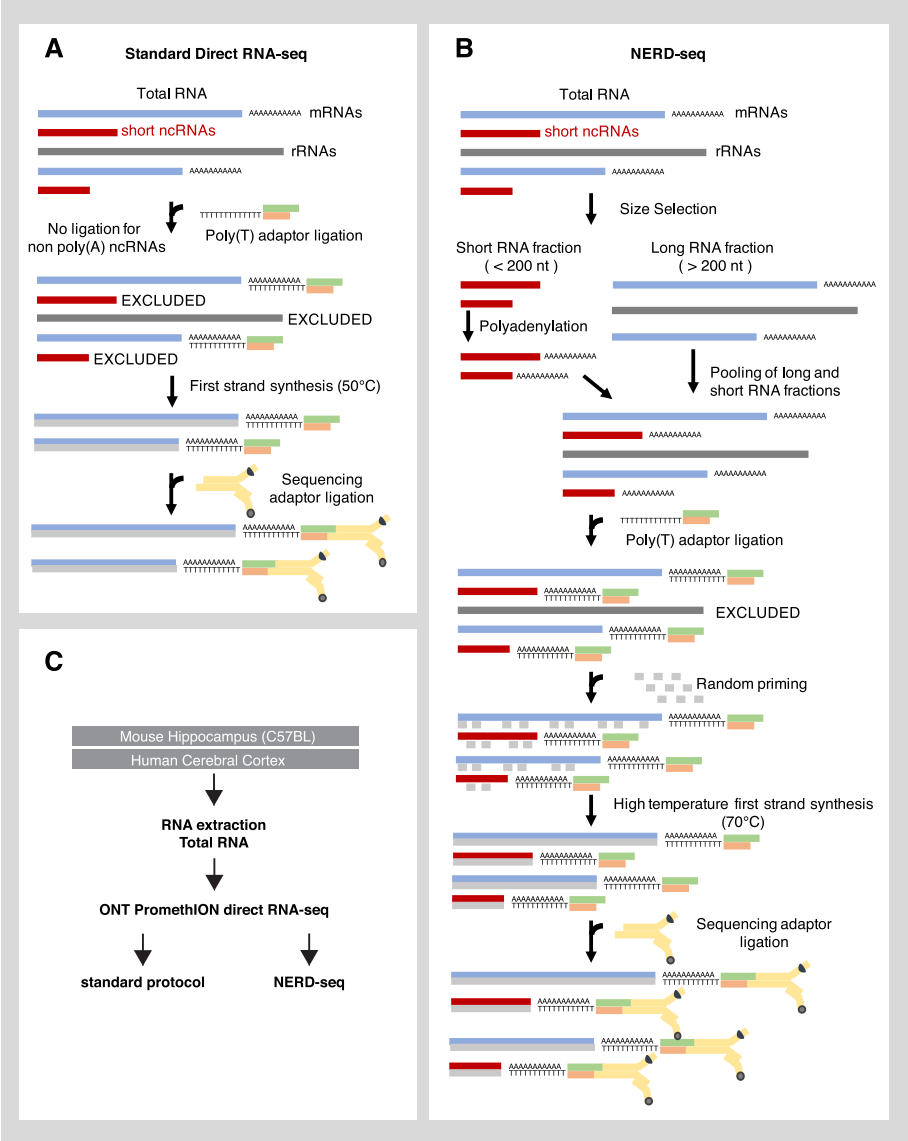
图1.NERD-seq方法概述
02 NERD-seq方法性能
为评估NERD-seq的潜力,研究团队利用从小鼠海马体提取的总RNA,对NERD-seq和标准直接RNA-seq测序结果进行了比较(图2)。结果显示,与标准直接RNA-seq方法相比,NERD-seq能够有效富集较短的非poly(A)RNA,可提供更多的信息,证明了该方法在神经组织RNA测序中的应用潜力。
研究团队通过NERD-seq技术对基因组元件进行了测序reads分析,发现与标准直接RNA-seq方法相比,NERD-seq在编码区和非编码区的reads分布有显著差异。标准直接RNA-seq主要关注编码区,尤其是已知外显子;而NERD-seq能够生成更多、具有更高覆盖度的非编码区reads。与标准方法相比,LINE是NERD-seq中代表性过剩的主要非编码元件之一。
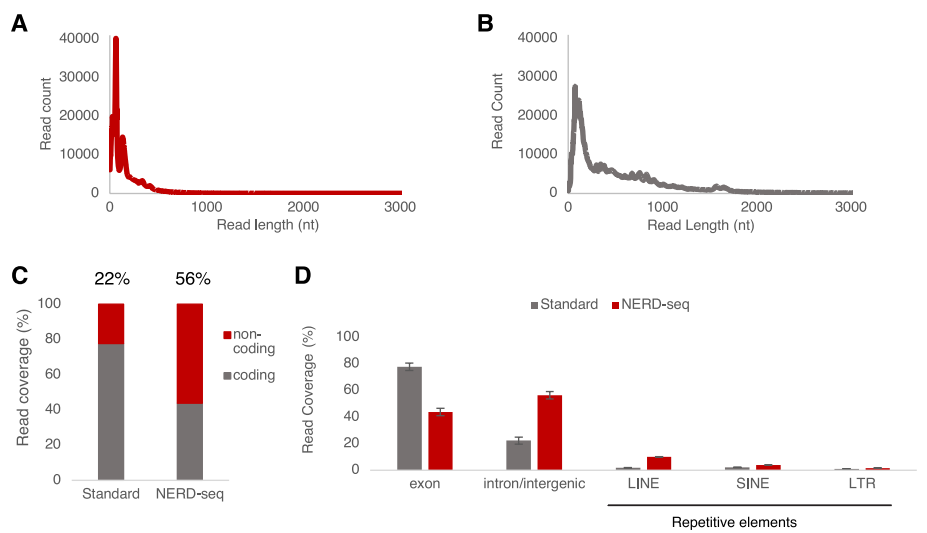
图2.NERD-seq能够为非编码基因组生成具有更高覆盖率的reads
除生成覆盖度更高的非编码基因组reads外,NERD-seq还能有效检测mRNA和poly(A) ncRNA。经验证,NERD-seq能够检测到转录起始位点(TSS)下游reads分布中峰值所记录的mRNA;在小鼠中NERD-seq成功检测到由SINE元件B2产生的全长poly(A) ncRNA(B2 RNA),并在人类大脑皮层组织中检测到丰富的、与人类B2 RNA同源的Alu SINE RNA。特别地,NERD-seq还能像标准方法一样分析mRNA的剪接模式。
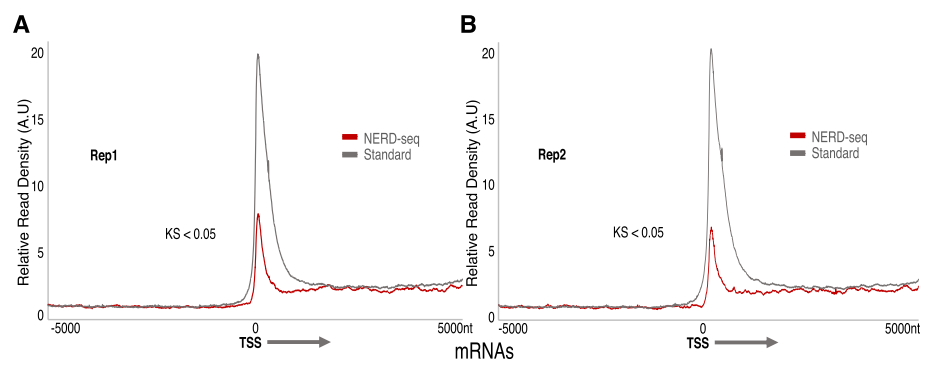
图3.NERD-seq可以可靠地检测mRNA
综上,NERD-seq技术在检测poly(A) ncRNA和分析mRNA剪接模式方面显示出与标准方法相似的性能,可有效检测到poly(A)转录物及其亚型。
03 NERD-seq可有效地检测各种类型的短非poly(A) ncRNA
为评估NERD-seq能否检测已知类型的非poly(A) ncRNA,研究团队生成了四类ncRNA(snoRNA、snRNA、scRNAs和srpRNA)的TSS周围相对reads密度分布图,并与标准方法和外部直接RNA-seq数据进行比较(图4)。结果显示,只有NERD-seq为所有四个类型ncRNA生成了稳健的分布。
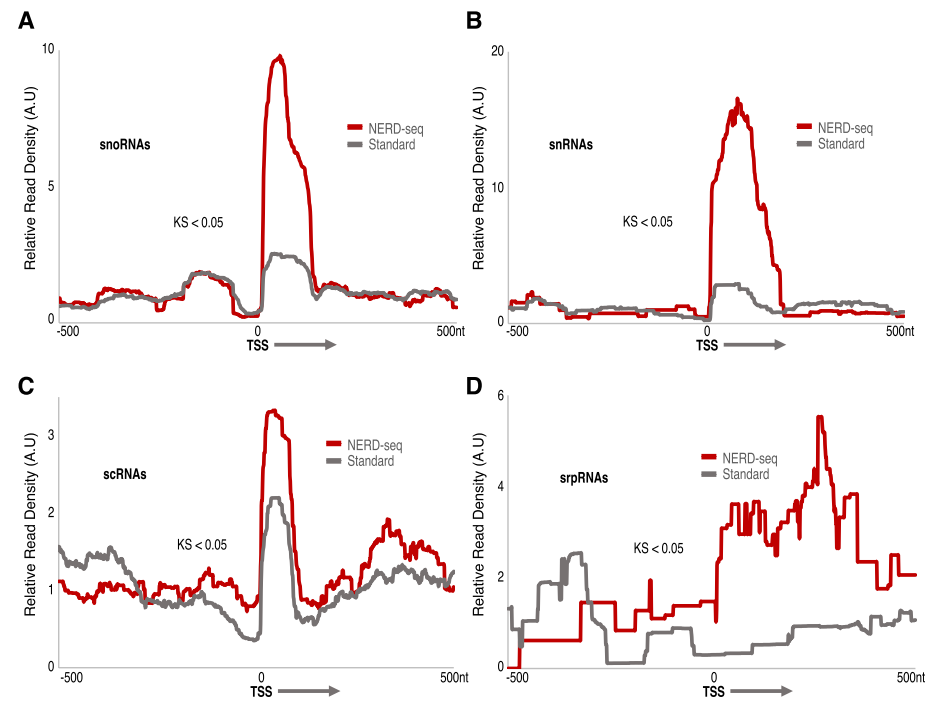
图4.NERD-seq可检测snoRNA、snRNA、scRNA 和srpRNA
研究团队还评估了NERD-seq对另一种重要的非poly(A)ncRNA——tRNA的检测性能。结果显示,NERD-seq能够在tRNA的TSS周围产生稳健的分布,其检测到的tRNA数量远超标准RNA检测方法;并能够产生与宏基因组数据类似的谱图。上述结果表明,NERD-seq可有效扩展直接RNA-seq功能,检测到标准RNA-seq可能遗漏或测序数量不足的多类短非poly(A)ncRNA。
04 NERD-seq将表观转录组学特征的研究扩展到更多的ncRNA类别
来自LINE的ncRNA在预防神经退行性疾病中具有重要作用,研究团队将通过NERD-seq和标准方法产生的reads映射到LINE元基因上,以识别LINE元件中的热点。与标准文库相比,NERD-seq数据中映射到LINE元件的reads更加丰富;在NERD-seq检测到的所有主要LINE元件中,来自LINE L1家族的reads覆盖率最高。这些结果表明,NERD-seq技术能够检测到源自LINE元件内部的ncRNA,并能从测序reads中提取更多无法通过标准方法获得的信息。
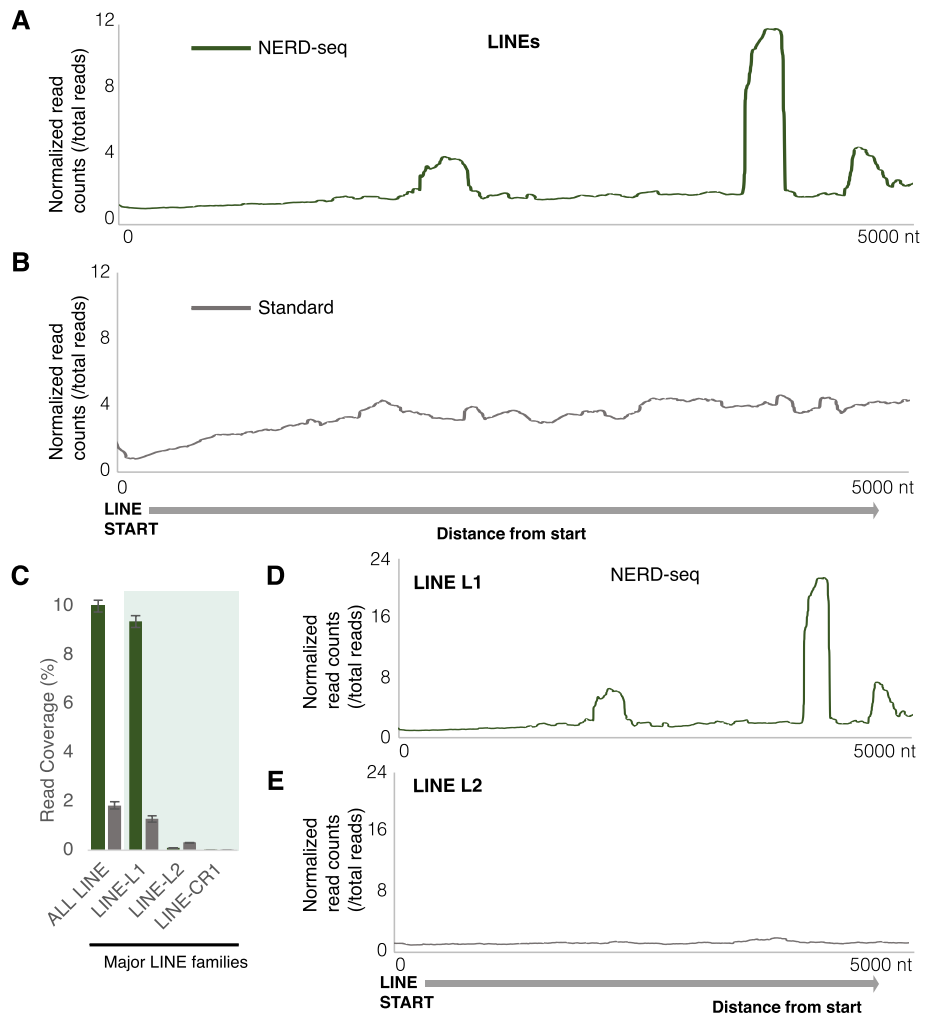
图5.NERD-seq检测LINE L1相关的ncRNA
为评估NERD-seq生成的数据在检测RNA修饰方面的有效性,研究团队使用来自合成构建体的人类7SK基因进行体外转录(IVT),并将其与通过标准和NERD-seq方法从人类大脑皮层(5个供体)测序的总RNA进行比较。与标准测序方法相比,NERD-seq无需使用独特的adapter便能捕获更多的7SK reads 以及其他多种RNA分子。
此外,研究团队还观察了GRCh38参考基因组中的rn7SK基因组区域,确认了尿嘧啶250处的假尿苷位点存在强烈的T/C错配;并发现了可能的肌苷位点和APOBEC底物相关的错配位点。
上述结果表明,NERD-seq方案在分析RNA修饰和表观转录组特征方面具有潜在的优势,在不需要特殊adapter 的情况下能够捕获更多的RNA分子。
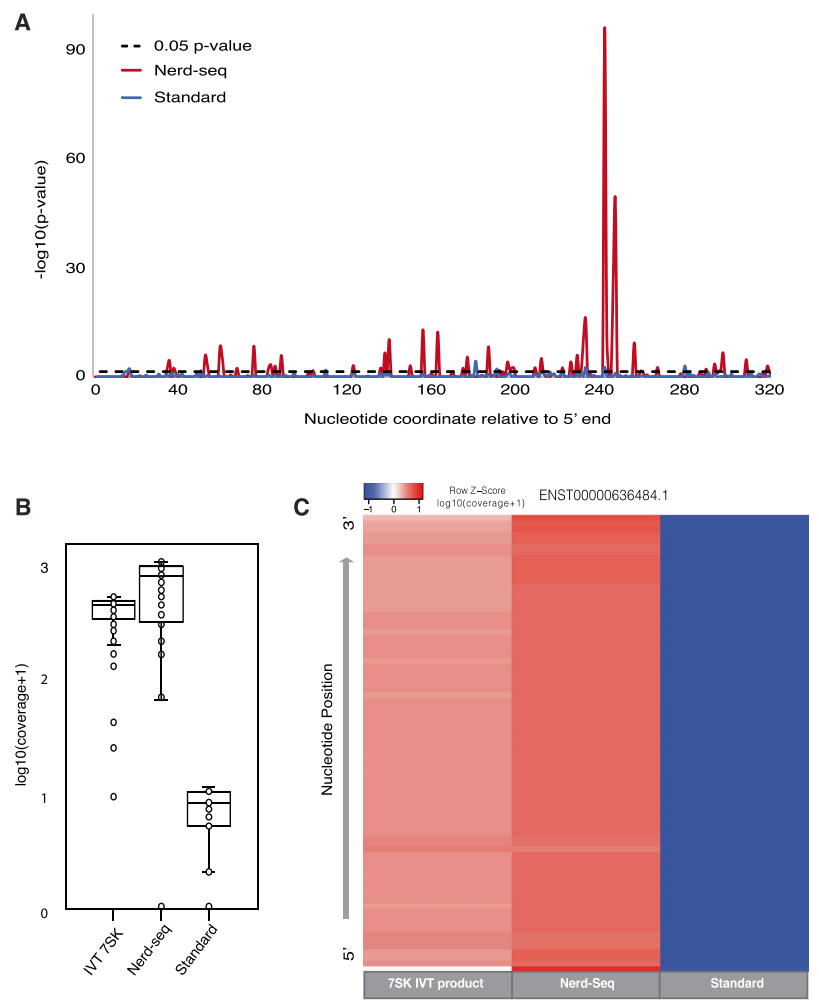
图6. NERD-seq可检测7SK RNA中的RNA修饰
综上所述,研究团队提出了一种名为NERD-seq的非编码RNA直接测序方法,该方法改进了标准的直接RNA测序文库技术,能够同时富集和捕获多种类型的ncRNA和mRNA。通过量化重要ncRNA类型的相对富集,研究团队证明了NERD-seq的效用,并评估了snRNA的表观转录组标记。总的来说,NERD-seq能够促进许多重要的ncRNAs相关研究,将其与mRNA的标准分析相结合可使转录组和表观转录组分析更加全面。
论文原文:
Saville L, Wu L, Habtewold J, et al. NERD-seq: a novel approach of Nanopore direct RNA sequencing that expands representation of non-coding RNAs. Genome Biol. 2024;25(1):233. Published 2024 Aug 28. doi:10.1186/s13059-024-03375-8


