湖南大学史俊峰/曾湘祥《自然·通讯》:基于深度学习框架开发的广谱抗菌肽,有效应对耐药细菌感染
时间:2024-09-02 16:00:49 热度:37.1℃ 作者:网络
开发强效且广谱的抗菌肽(AMPs)可能有助于克服抗菌药物耐药性危机。我们开发了一个基于肽语言的深度生成框架(deepAMP),用于识别强效、广谱的AMPs。利用deepAMP减少抗菌耐药性并增强AMPs的膜破坏能力,湖南大学史俊峰/曾湘祥&克利夫兰诊所Feixiong Cheng在两轮设计中通过交叉优化验证,识别、合成并实验测试了18种T1-AMP(第一层级)和11种T2-AMP(第二层级)候选物。超过90%的设计的AMPs在抑制革兰氏阳性菌(如金黄色葡萄球菌)和革兰氏阴性菌(如肺炎克雷伯菌和铜绿假单胞菌)方面显示出比渗透素更好的效果。T2-9显示出最强的抗菌活性,与FDA批准的抗生素相当。该研究展示了三种AMPs(T1-2、T1-5和T2-10)与环丙沙星相比显著降低了对金黄色葡萄球菌的耐药性,并对感染铜绿假单胞菌的雌性小鼠皮肤伤口模型中的感染有效。总之,deepAMP加快了针对耐药细菌的有效、广谱AMPs的发现。该研究以题为“A Foundation Model Identifies Broad-Spectrum Antimicrobial Peptides against Drug-Resistant Bacterial Infection”的论文发表在《Nature Communications》上。

图1展示了deepAMP框架的工作流程,包括使用UniProt数据库进行预训练,构建AMPs训练对和Penetratin训练对数据集,以及通过退化工作流程来微调预训练模型。利用deepAMP-POM模型优化Penetratin,并通过deepAMP-predict对候选抗菌肽进行排名。从排名中选择了第一轮18个序列进行体外和体内实验验证。这一流程证明了deepAMP能够有效地从大量候选肽中识别出具有高抗菌活性和低溶血性的抗菌肽,为后续的实验验证提供了科学依据。
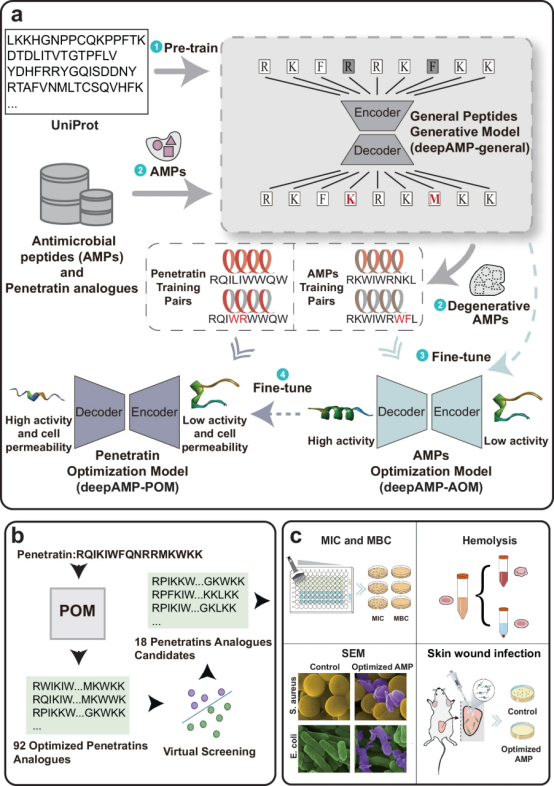
图1.通过深度学习框架deepAMP优化的抗菌肽的发现流程
【deepAMP优化性能的比较,包括与其他方法的迭代优化结果】
图2展示了deepAMP在优化抗菌肽方面的性能,比较了deepAMP-TOM(本研究中的模型)与Baseline-T、Random mutants、PepCVAE和HydrAMP等其他四种方法在一轮、两轮和三轮迭代后的优化结果。结果显示,除了deepAMP-TOM之外,所有方法在第一轮迭代后的优化结果均低于原始序列的评分。经过三轮迭代后,所有方法的平均评分均高于原始序列,但deepAMP-TOM的评分最优。此外,deepAMP-predict的评分结果显示,deepAMP-TOM在AMP优化方面具有最佳性能。在Pg-AMP1片段优化任务中,deepAMP-GOM在第三轮迭代时就超过了Baseline-G的健身分数,并且在五轮迭代后,改进了前100名候选肽的评分,表现优于现有方法,并达到了最高的0.594分。这些结果表明deepAMP在优化抗菌肽方面具有高效率、准确性,并能有效地识别具有广谱抗菌效果的潜在抗菌肽。
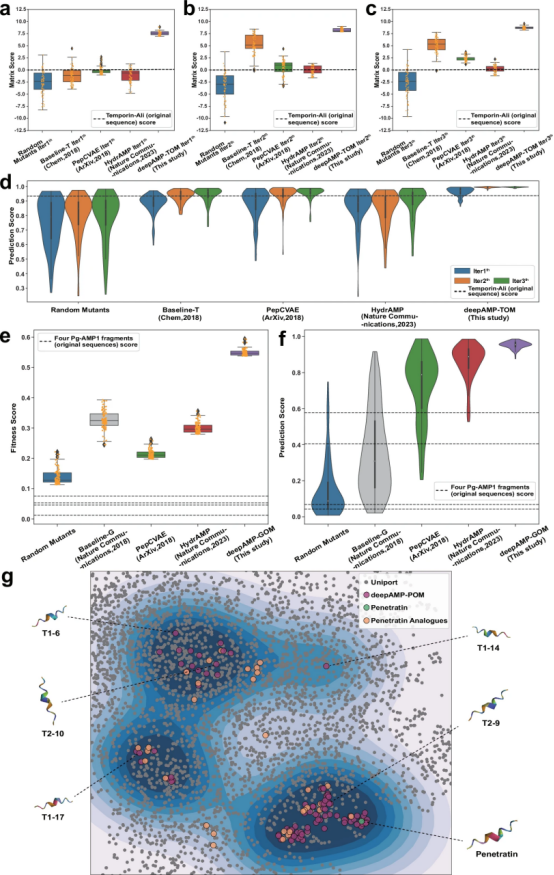
图2. deepAMP优化性能的比较,包括与其他方法的迭代优化结果
【候选抗菌肽的抗菌和溶血活性】
图3展示了deepAMP预测的候选抗菌肽的抗菌和溶血活性。图中描述了最小抑制浓度(MIC)和最小杀菌浓度(MBC)测试的方法,并展示了这些候选抗菌肽对多种细菌(包括大肠杆菌、金黄色葡萄球菌、肺炎克雷伯菌、铜绿假单胞菌和耐甲氧西林金黄色葡萄球菌)的MIC和MBC值。实验结果表明,与原始的抗菌肽Penetratin相比,deepAMP识别的大多数候选抗菌肽展现出更低的MIC值,显示出更强的抗菌活性。特别是T1-2和T1-5在抑制金黄色葡萄球菌方面的效果与FDA批准的抗生素左氧氟沙星相当。此外,还展示了候选抗菌肽的溶血活性测试结果,其中T1-6、T1-14、T2-9和T2-10不仅具有高抗菌活性,而且具有较低的红细胞溶血率(小于20%),表明它们具有良好的生物相容性。
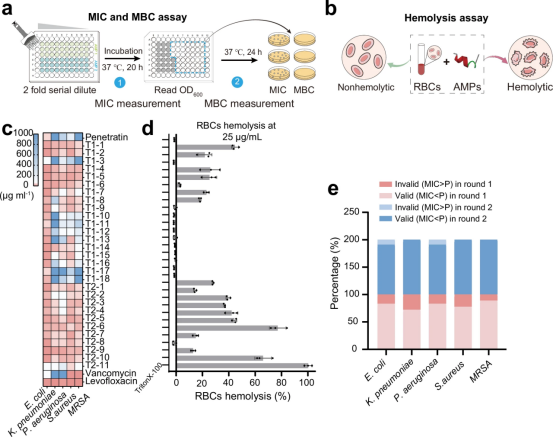
图3. 候选抗菌肽的抗菌和溶血活性
【候选抗菌肽对细菌膜的影响】
图4展示了候选抗菌肽对细菌膜的影响。通过使用活细胞染料Hoechst和死细胞染料PI进行染色,研究者观察了大肠杆菌和金黄色葡萄球菌在经T1-2和T2-10处理后的细胞膜完整性。结果显示,经过抗菌肽处理的细菌发出红色荧光,表明抗菌肽能够诱导细菌膜上形成孔洞,使PI染料进入细菌内部。此外,通过流式细胞仪检测,进一步证实了抗菌肽对细菌膜的破坏作用。这些结果表明,deepAMP设计的候选抗菌肽能够通过破坏细菌的细胞膜来发挥抗菌作用,从而有效地抑制细菌的生长。
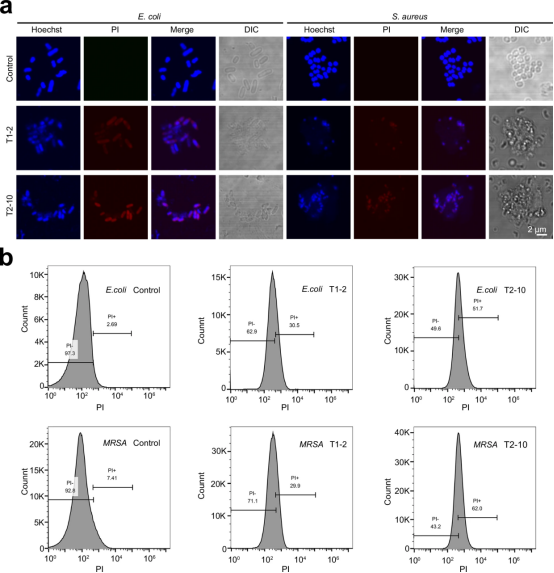
图4. 候选抗菌肽对细菌膜的影响
【候选抗菌肽的作用机制研究和耐药性发展】
图5展示了deepAMP设计的候选抗菌肽的作用机制研究和耐药性发展测试。通过荧光染色和流式细胞仪检测了抗菌肽对细菌细胞膜的破坏作用,以及通过膜电位敏感染料DiSC3(5)和NPN荧光探针测定了抗菌肽对细菌外膜的渗透能力。实验结果表明,这些抗菌肽能够显著增加DiSC3(5)和NPN的荧光强度,显示出它们能够破坏细菌的细胞膜和外膜。此外,通过连续传代实验评估了抗菌肽诱导细菌耐药性的能力,结果显示T1-2、T1-5和T2-10在25次传代后并未诱导出耐药性,而对照组的抗生素环丙沙星在10次传代后就开始出现耐药性。这些结果表明,deepAMP设计的抗菌肽不仅能够有效破坏细菌的细胞膜,而且在抑制细菌耐药性发展方面具有潜在的优势。
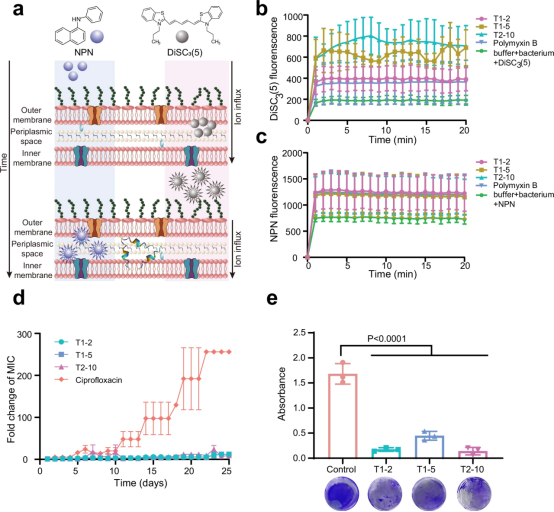
图5. 候选抗菌肽的作用机制研究和耐药性发展
【体内抗感染活性评估的实验设计和结果】
图6展示了deepAMP设计的候选抗菌肽在体内对抗感染的效果。通过在小鼠皮肤伤口感染模型中测试T1-2、T1-5和T2-10三种抗菌肽,研究发现这些肽在治疗后24小时内显著减少了感染区域的细菌数量,与阳性对照药物左氧氟沙星的效果相当。此外,通过组织病理学检查(H&E染色)观察到,与PBS处理组相比,经抗菌肽处理的小鼠伤口区域炎症细胞浸润程度较低,表明这些肽具有较好的抗感染效果且对组织损伤较小。这些结果表明,deepAMP框架识别的抗菌肽在体内模型中对治疗由铜绿假单胞菌引起的皮肤感染具有显著效果,并且具有良好的生物相容性,为未来治疗耐药菌株提供了有希望的候选物质。
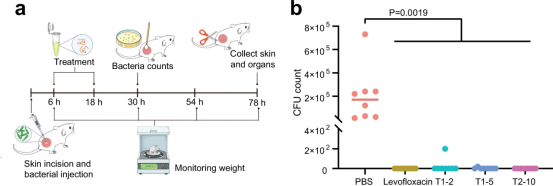
图6. 体内抗感染活性评估的实验设计和结果
【小结】
该研究开发了一个基于肽语言模型的深度生成框架(deepAMP),用于识别有效、广谱的抗菌肽(AMPs),以应对日益严重的抗生素耐药性危机。通过两轮设计和交叉优化验证,研究团队成功识别、合成并实验测试了18种Tier 1和11种Tier 2候选抗菌肽。这些设计的AMPs在抑制革兰氏阳性(如金黄色葡萄球菌)和革兰氏阴性细菌(如肺炎克雷伯菌和铜绿假单胞菌)方面表现出比渗透素更好的效果,其中T2-9显示出与FDA批准的抗生素相当的最强抗菌活性。此外,三种AMPs(T1-2、T1-5和T2-10)显著降低了对金黄色葡萄球菌的耐药性,并在感染铜绿假单胞菌的雌性小鼠皮肤伤口模型中显示出良好的效果。总之,deepAMP加速了有效、广谱抗菌肽的发现,这些抗菌肽有望对抗耐药细菌。
原文链接:


