Nat Genet:新单倍型定相方法SHAPEIT5,可在大型WGS/WES数据集中精确鉴定罕见变异
时间:2023-07-22 23:19:30 热度:37.1℃ 作者:网络
目前的遗传关联研究越来越多地基于全基因组或全外显子组测序(WGS/WES)。与之前基于单核苷酸多态性(SNP)阵列的研究相比,WGS和WES数据可以识别罕见变异,系统地表征它们对各种性状和疾病的贡献。在这种情况下,稀有变异的单倍型定相(haplotype phasing)涉及将每条染色体的遗传区域分为来自父方或母方的单倍型,这对识别复合杂合事件至关重要。因为复合杂合性是非近亲结婚个体罕见隐性疾病最常见的遗传模型之一。
近期,来自瑞士洛桑大学的研究团队在Nature Genetics上发表研究文章“Accurate rare variant phasing of whole-genome and whole-exome sequencing data in the UK Biobank”。该研究报道了一种新的单倍型定相方法SHAPEIT5,可以在大型WGS/WES数据集中精确鉴定罕见变异。通过对英国生物银行(UKB)全基因组和全外显子组测序数据的分析,研究团队证明SHAPEIT5可以提高基因型插补的准确性。

文章发表在Nature Genetics上
SHAPEIT5单倍型定相方法概述
据文章介绍,SHAPEIT5使用三种不同的定相模型对WGS或WES数据进行单倍型定相(图1),每个定相模型侧重于不同类型的变异:(1)使用SHAPEIT4模型对常见变异进行定相;(2)利用插补模型,将罕见变异分阶段植入所产生的单倍型;(3)采用合并启发模型对单例变异进行分阶段分析。SHAPEIT5对每个罕见杂合基因型进行定相,以少量信息丰富的单倍型为条件(图1b)。对于单例变异(次要等位基因计数(MAC)为1),SHAPEIT5使用另一种定相模型,该模型假设单例变异是最近的突变事件,并利用单倍型之间的IBD(Identical By Descent)共享模式进行推断(图1c)。
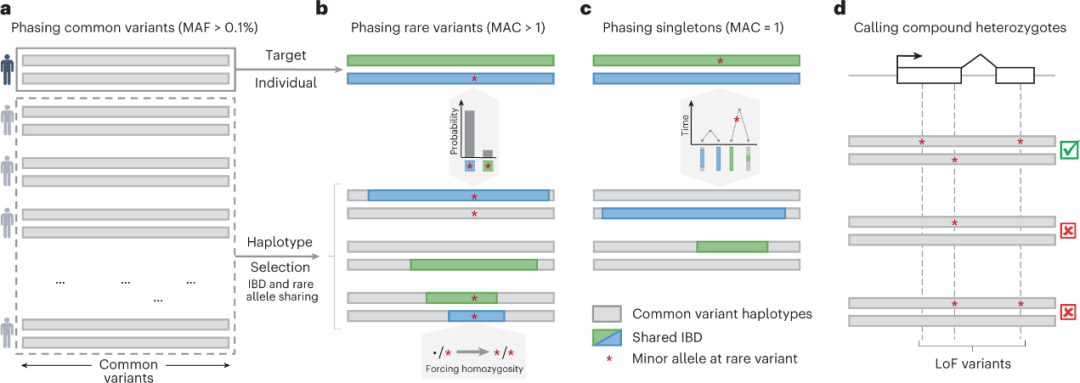
图1. SHAPEIT5的基本原理。
评估SHAPEIT5在UKB数据中的性能
为了评估定相性能,研究人员使用了147754个WGS数据和447470个WES数据,这些数据里包含了部分家系信息。研究发现,SHAPEIT5和Beagle v.5之间存在细微差别(图2)。当仅考虑Axiom阵列位点时,使用SHAPEIT5具有较低的转换错误率(SER)。
为了获取更可靠的数据,研究人员对次要等位基因计数(MAC)bin内的SER进行分层,以关注罕见变异。根据变异频率将杂合基因型分配到不同的MAC bin,并在每个MAC bin中计算它们被正确定相的分数。结果显示,SHAPEIT5在WGS和WES数据集中具有比Beagle v.5.4更高的准确度以对罕见变异进行定相(图2)。
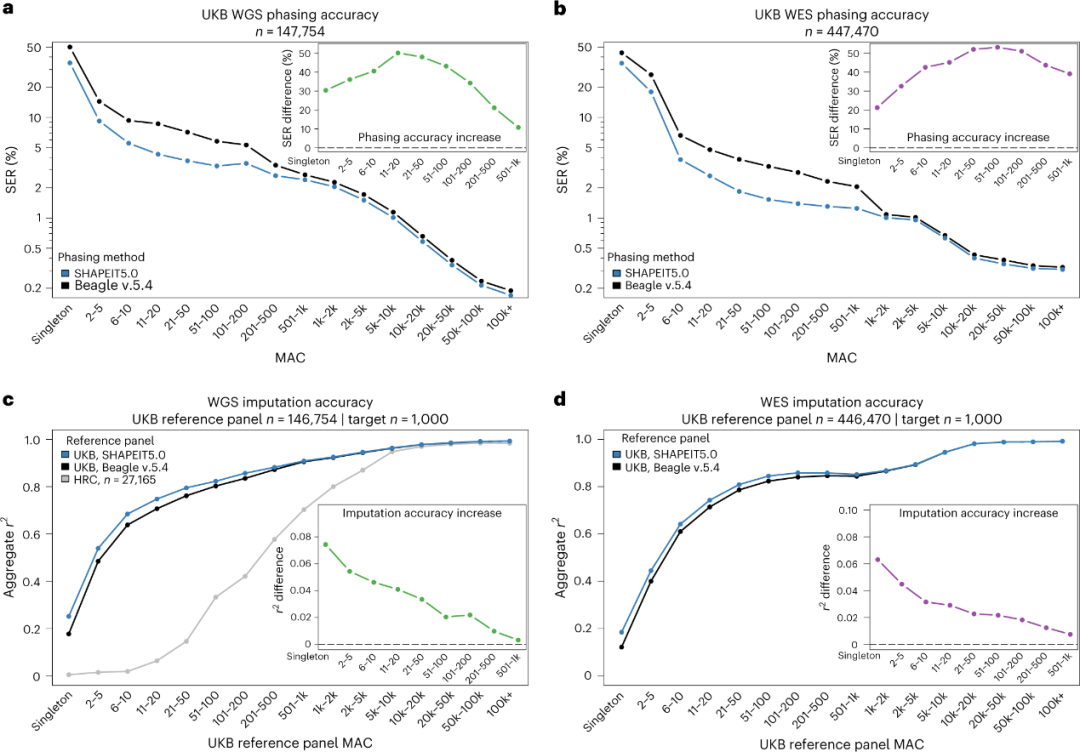
图2. SHAPEIT5在UKB数据中的分相性能。
SHAPEIT5可对LoF复合杂合子进行鉴定
当基因的两个拷贝都含有至少一种杂合变异时,被称为复合杂合突变。复合杂合性通常在LoF变异的背景下进行研究,预计这些变异会对基因产生高度有害的影响,相当于纯合基因敲除。研究人员在检测的17689个蛋白质编码基因中,发现2150个(12%)基因至少有一个个体具有两个或多个LoF变异。从这些基因中,研究发现549个(26%)基因与一个或多个具有复合杂合LoF变异的个体相关(图3a)。当仅考虑高置信度单倍型调用时,SHAPEIT5仍然识别出完整数据集中识别的80%基因和79%的复合杂合事件,表明这些主要依赖于高置信度单倍型调用。同时,研究发现,与具有两个或多个LoF变体的2150个基因相比,549个复合杂合基因在几个已知必需基因列表中高度缺失(图3b)。相反,复合杂合基因在非必需和纯合LoF耐受基因列表中丰富(图3c)。总体而言,SHAPEIT5对罕见变异的准确定相能够以高置信度筛选UKB队列中的复合杂合事件,揭示LoF复合杂合事件在必需基因中面临强大的选择压力,正如其高负面影响所预期的那样。
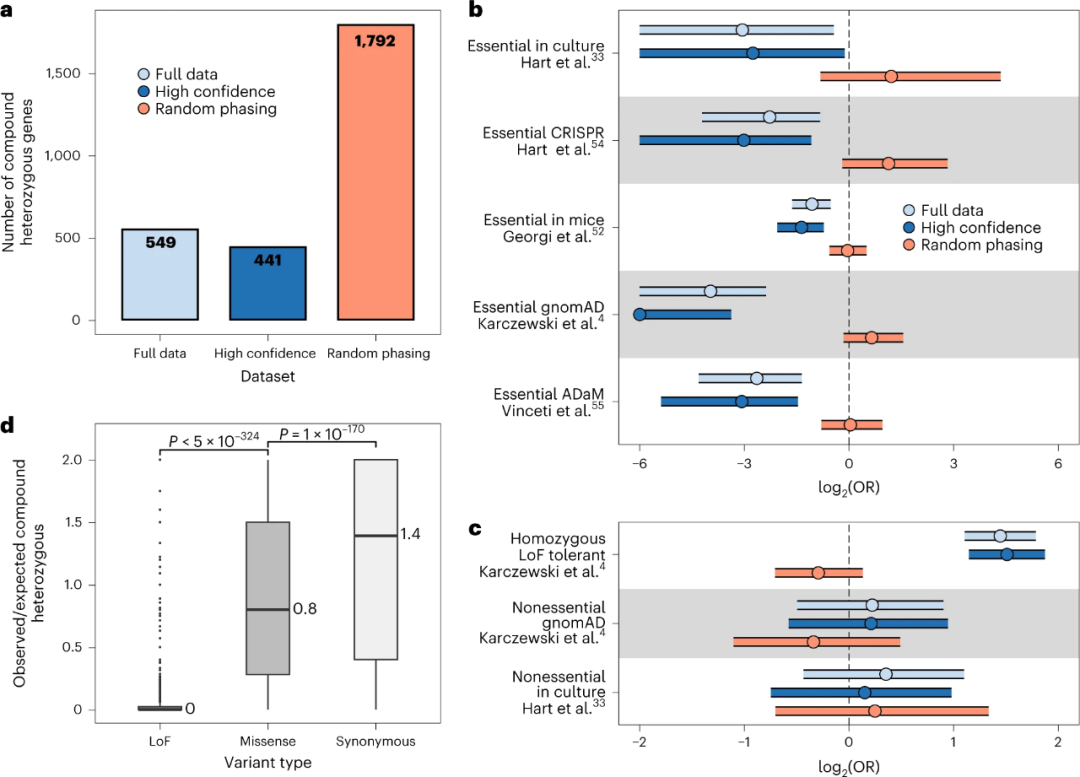
图3. 使用SHAPEIT5定相的UKB WES数据中的复合杂合子鉴定。
结语
研究团队推出了SHAPEIT5,这是一种用于大型测序数据集罕见变异的定相工具。对UKB等大型测序研究中的罕见变异定相有可能解锁许多应用和分析。首先,可以筛选其他类型的功能变体的复合杂合效应。其次,阶段信息可以包含在罕见变异负荷测试方法中,该方法通常只考虑两种单倍型的混合。第三,使用精确定相的参考组可以对来自同一群体的任何新测序基因组进行高精度的极其罕见的变体定相,甚至在某种程度上是单例。这有利于诊断由复合杂合效应引起的罕见和严重疾病,例如英格兰基因组学数据集,其中可以通过合并相位信息来提高诊断率。
参考资料:
Hofmeister, R.J., Ribeiro, D.M., Rubinacci, S. et al. Accurate rare variant phasing of whole-genome and whole-exome sequencing data in the UK Biobank. Nat Genet (2023). https://doi.org/10.1038/s41588-023-01415-w


